重磅来袭:T2T基因组(动植物基因组完成图)全国招募
T2T(Telomere-to-telomere) 或Gapless 基因组是指基因组准确性高、连续性高、完整性高的端粒到端粒的高质量基因组。三代测序技术的发展,特别是高连续性的ONT ultra-long 测序和高准确性的HIFI测序强强联合,克服了着丝粒或高重复区域的组装困难问题,染色体的连续性和完整性大大提高,为T2T基因组的组装奠定了基础,使基因组组装进入近0 gap时代。
T2T基因组的组装,有助于对基因组中高重复序列区域或高重复结构进行深入研究,为着丝粒区域或未知高重复区域的变异特征的研究提供契机。
AG代理基因长期致力于提高三代ONT测序数据的产量和质量的研发,关注ONT ultra-long对基因组组装质量的提升,是国内少数能稳定产出高质量ONT ultra-long 数据的服务商之一。目前,已实现ONT ultra-long单张芯片产出超20G的测序reads N50>100Kb的数据;已经完成多个基因组的ultra-long reads组装。
现推出ONT ultra-long结合HIFI的高性价比的T2T基因组测序和组装策略,并限量推出 “ T2T基因组测序半价,组装免费 ” 的优惠活动!
AG代理基因特别推出:
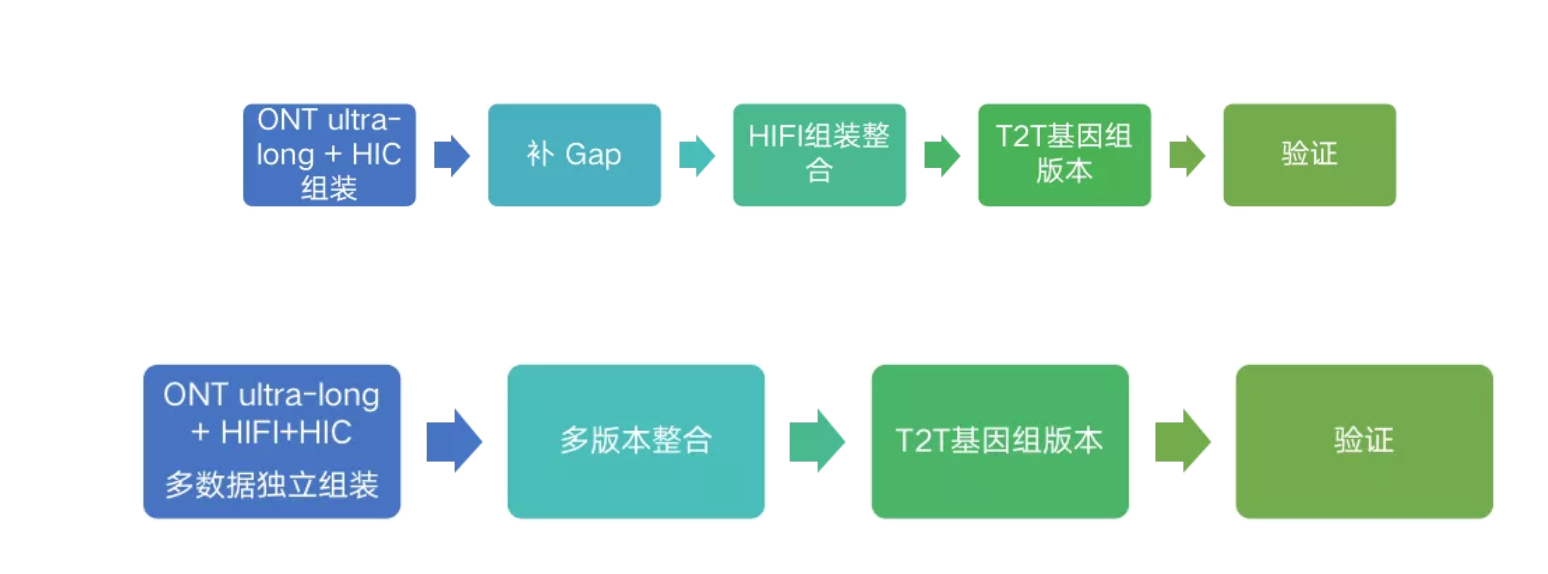
最强T2T测序策略
ONT ultra-long (N50:100kb) 50-100x+HIFI 100x
最强T2T组装策略
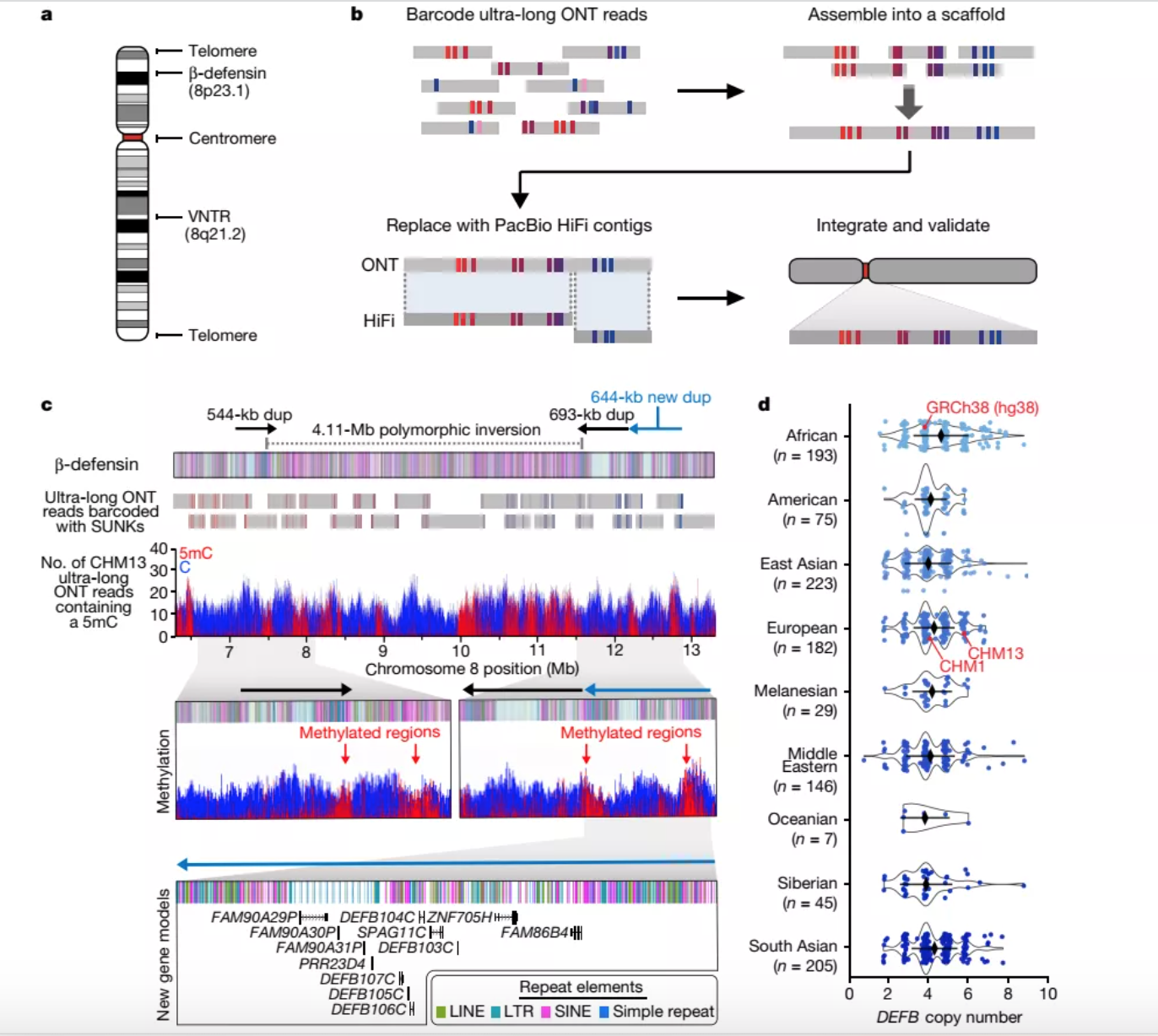
参考案例1:The structure, function and evolution of acomplete human chromosome 8
期刊: Nature (IF = 49.962 )
组装策略: ONT ultra-long (20x)+HiFi (32.4x)
研究结果:
首次使用互补的长读测序技术完成了对于人类8号染色体的线性组装和解析,
解析了5个以前长期存在的缺口序列。
证实了二倍体人类基因组中着丝粒的整体结构和甲基化模式。
填补了对于染色体中着丝粒重复区域的认识。
参考案例2:High-quality Arabidopsis thaliana genome assembly with Nanopore and HiFi long reads
期刊: Genomics Proteomics Bioinformatics (IF= 7.691)
组装策略: ONT ultra-long > 50kb (177x)+ HiFi (153x)
研究结果:
实现了仅剩两个缺口的高质量拟南芥基因组Col-XJTU。
碱基准确性和结构准确性均高于目前的参考基因组TAIR10.1,QV>60。
展示了着丝粒区域的多样性,研究了CENH3 富集区域的甲基化模式。
为了解拟南芥自然自交系中着丝粒多态性、遗传和表观遗传的全球模式提供有价值的参考。

 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



