文献解读 | 人类Y染色体完整序列公布!
研究背景
2022年12月1日,题为“The complete sequence of a human Y chromosome”的研究在预印本平台bioRxiv上发表。众所周知,人类Y染色体由于其复杂的重复结构(包括长回文、串联重复和片段重复)而难以测序和组装。GRCh38是第一次包含了着丝粒序列的人类参考基因组版本,但仍然有超过一半的Y染色体缺失。该研究中,T2T联盟提供了来自HG002基因组(T2T-Y)的人类Y染色体的完整的62460029个碱基对序列,该版本纠正了GRCh38-Y中的多处错误,同时还增加了3000多万个碱基对,揭示了TSPY、DAZ和RBMY完整的扩增子区结构,另外,还补充了42个额外的蛋白质编码基因以及提出了异染色质区域Yq12中人类卫星1和3区块的交替模式。最后,该研究将T2T-Y与CHM13版本基因组相结合,获得了一些群体变异、临床变异和功能基因组学数据,来获得完整和全面的人类22+XY染色体的参考序列。
文章标题:
The complete sequence of a human Y chromosome
文章链接:
The complete sequence of a human Y chromosome | bioRxiv
主要研究结果
1、 T2T-Y的组装、验证和注释
基于PacBio HIFI 、ONT和illumina 三种测序技术获得的reads,遵循先前T2T-CHM13组装所用的策略,该研究对Y染色体进行了组装。最终组装得到的T2T-Y大小为62460029bp,没有gap或者模型序列,且估计的误差率为每10Mb少于1个误差。该研究团队将组装得到的T2T-Y(源自HG002)与先前的T2T-CHM13v1.1结合创建了新的Y-bearing 参考组装,并在此次研究中将其称为T2T-CHM13+Y。
该研究还进行了全面的重复注释,在T2T-Y中识别到了29个先前未知的重复,在GRCh38-Y中染色体只有30.58%被注释为重复序列,而T2T-Y中则有84.86%的重复。T2T-Y的重复注释还改进了HG002和CHM13中的ChrX注释,特别是在PAR区,每个ChrX增加了约33kb的卫星注释。文章对T2T-Y进行注释,共注释到693个基因和888个转录本,其中107个基因(493个转录本)被预测为蛋白质编码基因。与GRCh38-Y比较发现,共有6个基因的注释存在差异:其中4个基因在两个基因组中都得到了正确的注释,但是由于推测的序列水平差异导致分配到了不同的同源基因名称,另外两个差异注释基因是T2T-Y中缺失的TSPY假基因,这些假基因靠近GRCh38中的TSPY组装gap,因此判断可能是GRCh38-Y中该区域装配错误导致的错误注释。另外,除了包含GRCh38-Y中注释的所有基因外,T2T-Y还包含另外110个基因,其中42个被预测为蛋白质编码基因,而且这些PCG中的大多数是TSPY的额外copy,这填补了GRCh38-Y中的相应空白。
2、 与GRCh38-Y的比较
之前的研究已经明确,GRCh38-Y由两个序列组成,较长序列中超过一半(30.8Mb)代表了异染色质区块和亚端粒或者卫星重复序列的gap,较短序列(37.2kb)在组装时也只是被分配到了ChrY上但没有被定位;此外,GRCh38-Y中的PAR1(2.77Mb)和PAR2(329.5kb)并不是从头组装而是从ChrX复制而来,且着丝粒也是由227kb的模型序列来表示。该研究则将T2T-Y和GRCh38-Y直接进行序列比较,发现在对齐区域中排除gap外具有约99.8%的平均一致性,但仍存在多种结构差异如GRCh38-Y的着丝粒模型区。T2T-Y揭示了长臂异染色质区先前没有表征的约30Mb序列,为了进一步了解T2T-Y和GRCh38-Y之间的基因组差异,作者还分别确定了各自的Y染色体单倍群,它们由积累在雄性特异区域MSY非结合部分的突变决定;通过建树和数据库比对,该团队确定了T2T-Y和GRCh38-Y的单倍群分别为J-L816(J1)和R-L20(R1b),这些单倍群分别在阿什肯纳兹犹太人和欧洲人中最常见,与这些基因组已经确定的祖先相一致(表1,图1)。
表1 GRCh38-Y和T2T-Y的比较


图1 完整的Y染色体结构
a:GRCh38-Y和T2T-Y之间的直接比较;b:T2T-Y着丝粒的结构;
c:GRCh38-Y和T2T-Y中P1-P3区域的回文结构比较;
d:左上图显示了通过DNA染料(DAPI)标记的所有染色体;右上图显示用FISH探针标记的ChrY;中间图表示每个DYZ2/DYZ1重复单元与其共有序列的一致性百分比;下图显示DYZ2和DYZ1重复单元的GC序列组成百分比以及DYZ2中一个古老AluY片段的位置。
3、 着丝粒
正常的人类着丝粒有很多富含AT的α卫星家族,且通常会排列成更高阶的重复结构,其周围环绕着更多分散的α和其他卫星序列。以前的T2T-CHM13组装包括所有的人类染色体完全组装的着丝粒,除了ChrY。此外,由于其相对较短的长度,GRCh38 RP11 ChrY着丝粒先前的组装是基于RPCl-11 BAC文库的ONT测序数据完成。在该研究中,作者表征了T2T-Y着丝粒区域(J1单倍群)的序列,并与RP11(R1b单倍群)进行了比较:注释了T2T-Y中366kb的α卫星,其跨越了317kb的DYZ3 HOR阵列,尽管HOR阵列中的各个单元高度相似,但与RP11相比,全长重复单元共识别出了3种HOR亚型和一个不同的组织。另外,T2T-Y着丝粒阵列的大部分由具有一个36-mer小扩展的34-mers组成,其具有更长的HOR变异,但该情况在RP11中并不存在,取而代之的是一个包含内部重复的35-mer(图1b)。
4、 q臂异染色质的组成
人类Y染色体在q臂的远端(Yq12)包含一个大的异色区,几乎完全由两个分散的卫星序列(DYZ1和DYZ2)组成,GRCh38-Y中最大的一个缺口就位于Yq12,其只有极小的DYZ1和DYZ2代表性,该研究则揭示了单碱基分辨率下Yq12区域的详细结构,添加了超过20Mb的DYZ1和14Mb的DYZ2;在大约15%的Strand-seq文库中可以观察到,完整的Yq12组装也使得姐妹染色单体交换能够在这个异染色质区域内被定位。
5、 人类序列是基因组数据库中常见的污染物
人类DNA序列有时会作为污染物出现在其他物种得基因组中。在微生物研究中,人类参考序列被用于筛选污染的人类DNA,然而,由于当前参考序列的不完整性,一些人类片段被遗漏并错误地注释为细菌蛋白,导致公共数据库中存在数千个假蛋白。例如,该研究中对近5000个人类全基因组数据集进行分析发现,多个细菌物种与人类雄性之间存在意外的联系,因此作者使用完整的T2T-Y序列进一步探讨了污染问题,发现细菌数据库中的确存在人类ChrY污染片段(图2)。该团队预测这种污染问题尤其是高度重复序列污染类型包括来自所有人类染色体的序列,并扩展到所有序列数据库,包括非微生物基因组。
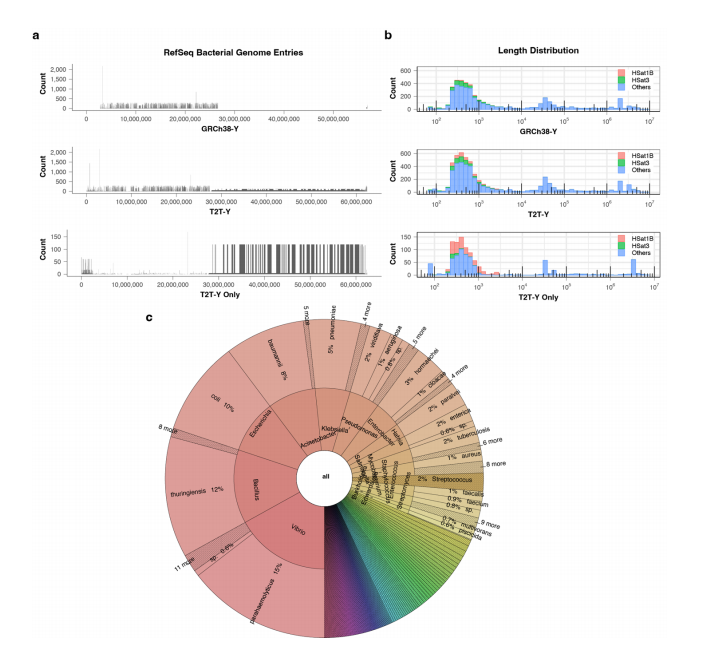
图2 细菌参考基因组中的人类污染物
总结
T2T-Y的组装挑战了之前CHM13基因组开发的组装方法,并促进了二倍体人类基因组组装新的自动化方法的开发。随着二倍体人类基因组的完整、准确和无缺口的组装成为常规,预计“参考基因组”将可能被称为简单的“基因组”。
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



