文献解读 | 使用超深宏基因组测序构建绵羊和山羊的肠道微生物Ref-宏基因组图谱
大多数关于反刍动物消化道的微生物组研究都集中在瘤胃微生物群上,仅进行了少数研究来调查反刍动物的肠道菌群,这限制了我们对这一重要成分的理解。本研究使用超深宏基因组测序表征了来自中国6个省的30只羊亚科(Caprinae)动物的肠道菌群。构建了包含5046个宏基因组组装基因组(MAGs)的Caprinae肠道微生物物种集,其中2530个属于未培养的候选物种。这些基因组在很大程度上扩展了Caprinae肠道中当前微生物的基因组库,丰富了对Caprinae肠道微生物群的分类、功能和代谢多样性的理解,为动物生产和动物健康的未来研究提供了基础。

文章标题:来自羊亚科肠道微生物群的5000多个宏基因组组装的微生物基因组集
发表期刊:Microbiology Spectrum(IF=9.043)
发表时间:2022.09.02
研究策略
1. 实验设计和样品收集
收集中国6个省(安徽、吉林、广西、山东,山西和西藏)的绵羊和山羊的30个粪便样品
2. DNA提取和文库制备、测序
Illumina NovaSeq平台(平均每个样品110.1 Gb)
3. 生物信息学分析
l 宏基因组组装和分箱
l MAGs的物种级聚类分析
l MAG的分类学、系统发育和功能分析
l Akkermansia, Treponema, Eggerthellales和Euryarchaeota的系统发育分析
主要研究结果
1. 来自Caprinae肠道的宏基因组组装基因组
对每个样品进行宏基因组组装和分箱,总共产生了5046个MAG,其中,1933MAG达到高质量标准,3113被鉴定为中等质量。MAGs的平均完整性和污染分别为86.3%和1.4%。值得注意的是,91.6%的MAG具有10 kb的组装长度(图1B),突出了这些MAG的高组装质量。MAG根据95%的ANI阈值分为3306种物种级基因组仓(SGBs)。这些SGB很好地代表了肠道中的物种(图1C)。SGB的系统进化树如图1D所示,细菌门主要包括厚壁菌,拟杆菌,变形杆菌,疣状杆菌等。优势科是Ruminococcuraceae,Lachnospiraceae和Rikenellaceae,只有14.3%的SGB可以被分类为属级。
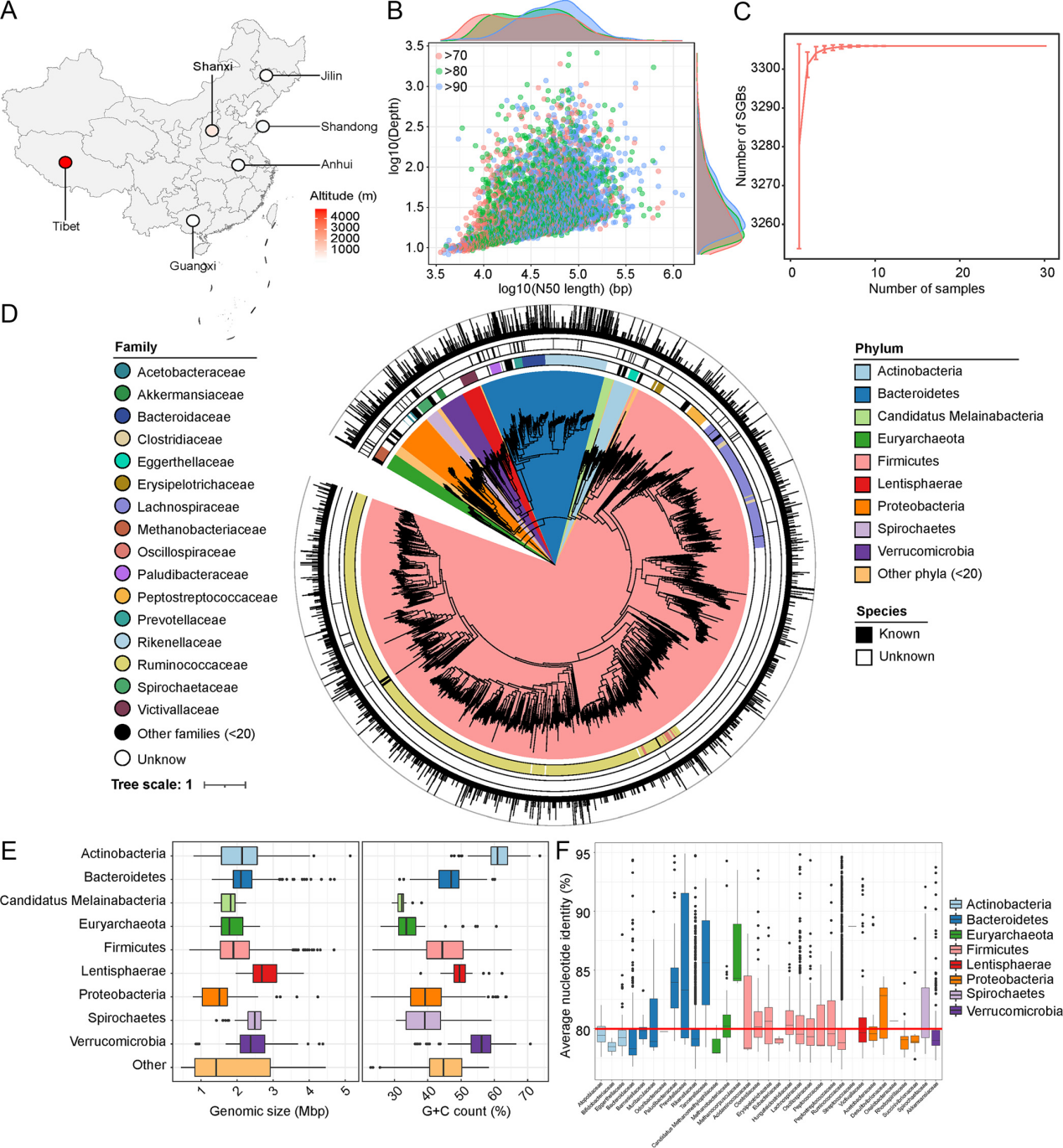
图1. 样本和MAG的统计数据
2. 哺乳动物相关微生物生命树的扩张
将3306个Caprinae肠道SGB与四个大规模可用数据集(反刍牛,RUG;人类胃肠道基因组,UHGG;非人类灵长类动物肠道微生物组,NHP;非冗余反刍动物胃肠道,RGIG)进行比较。拟杆菌,厚壁菌,放线菌和变形菌是几乎所有哺乳动物肠道中的优势物种。然而,Caprinae肠道具有显著的更高比例的厚壁菌成员和较低比例的拟杆菌、放线菌和变形菌成员(图2A)。Caprinae肠道中的物种与其他目录具有低重叠(图2B)。
Caprinae肠道有71种疣微菌,其中21种属于Akkermansia,这是人类和动物肠道中唯一发现的Verrucomicrobia属。研究发现21个Caprinae Akkermansia SGBs与已知的Akkermansia成员存在较远的进化距离(图2C)。肠道中Akkermansia群落的高度多样性可能表明Caprinae肠道是多种Akkermansia/Verrucomicrobia物种的进化库。此外,山羊亚科肠道也可能是蛋壳科、广古菌等的一个库(图2C)。这些发现和未来对这些未被充分代表的分类群的描述将极大地扩展知识与哺乳动物相关的微生物生命树。
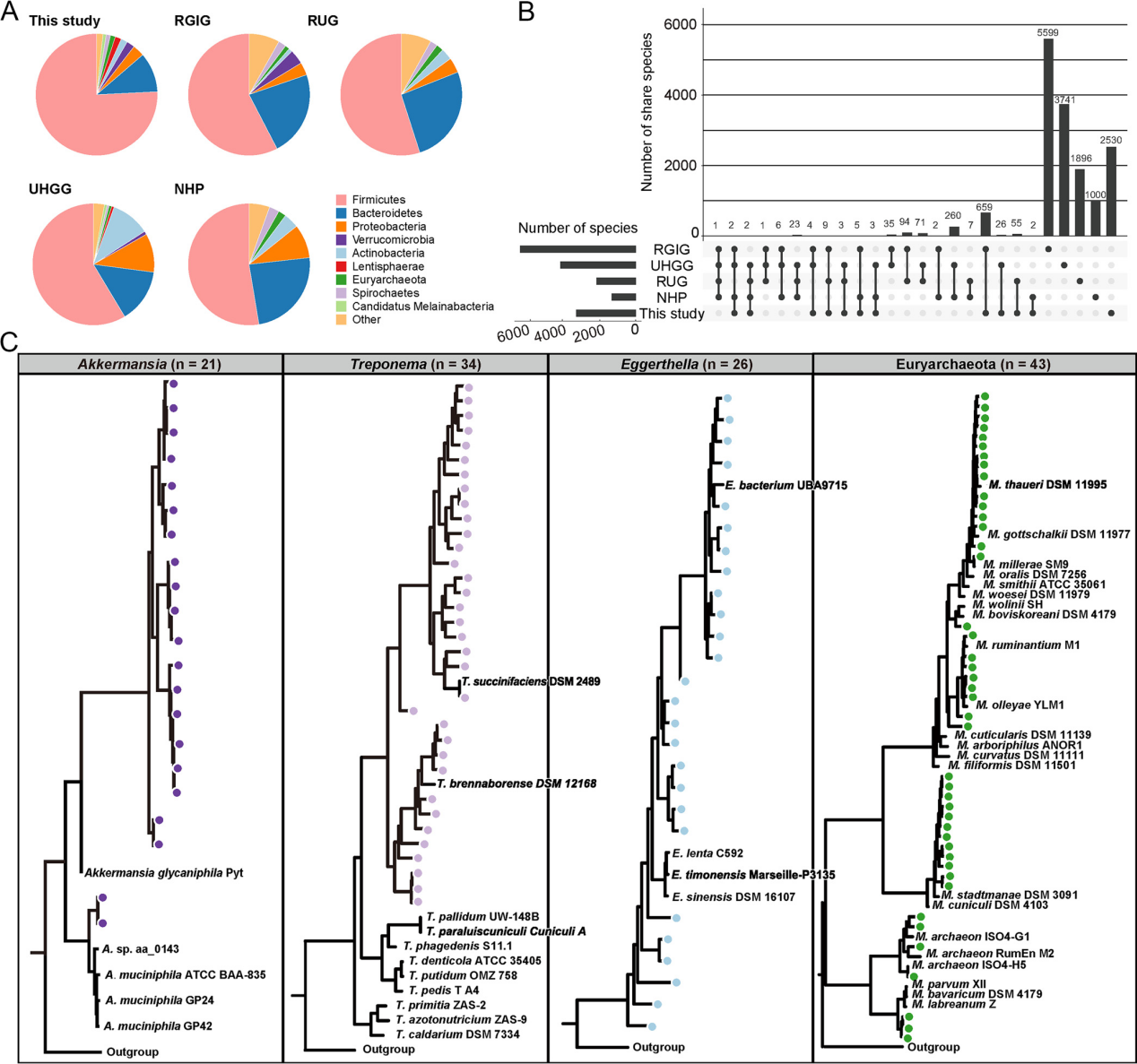
图2. 五种微生物基因组目录和山羊亚科特有分类群的系统发育学比较
3. Caprinae肠道菌群基因组的功能
KEGG数据库注释基因的比例在放线菌和变形菌中相对较高,但在疣菌中较低(图3A)。主坐标分析表明,MAGs的功能分布主要由它们的门级分层决定(图3B)。KEGG途径的比较揭示了MAGs的功能显著不同(图3C)。在KEGG模块级别,69个与代谢相关的模块在不同的门或家族中具有完整性差异(图3D)。Bacteroidaceae具有四个完整的核苷酸代谢模块,而这些模块在CAG-611和UBA1242家族中完全不存在,并在其他家族中以较低的水平出现(图3D)。表明Bacteroidaceae在核苷酸代谢方面的功能独特性。
从Caprinae肠道MAGs中鉴定出总共5586个次级代谢物生物合成基因簇,分为5大类和32种产物类型。从46个微生物菌株中鉴定出69个抗生素抗性基因,主要由变形菌,厚壁菌和拟杆菌成员携带,少量也由疣菌和放线菌携带。鉴定了来自17个MAGs的302个毒力基因。大多数含有毒力基因的MAGs属于变形杆菌,表明该门可能包含更多潜在的致病菌。
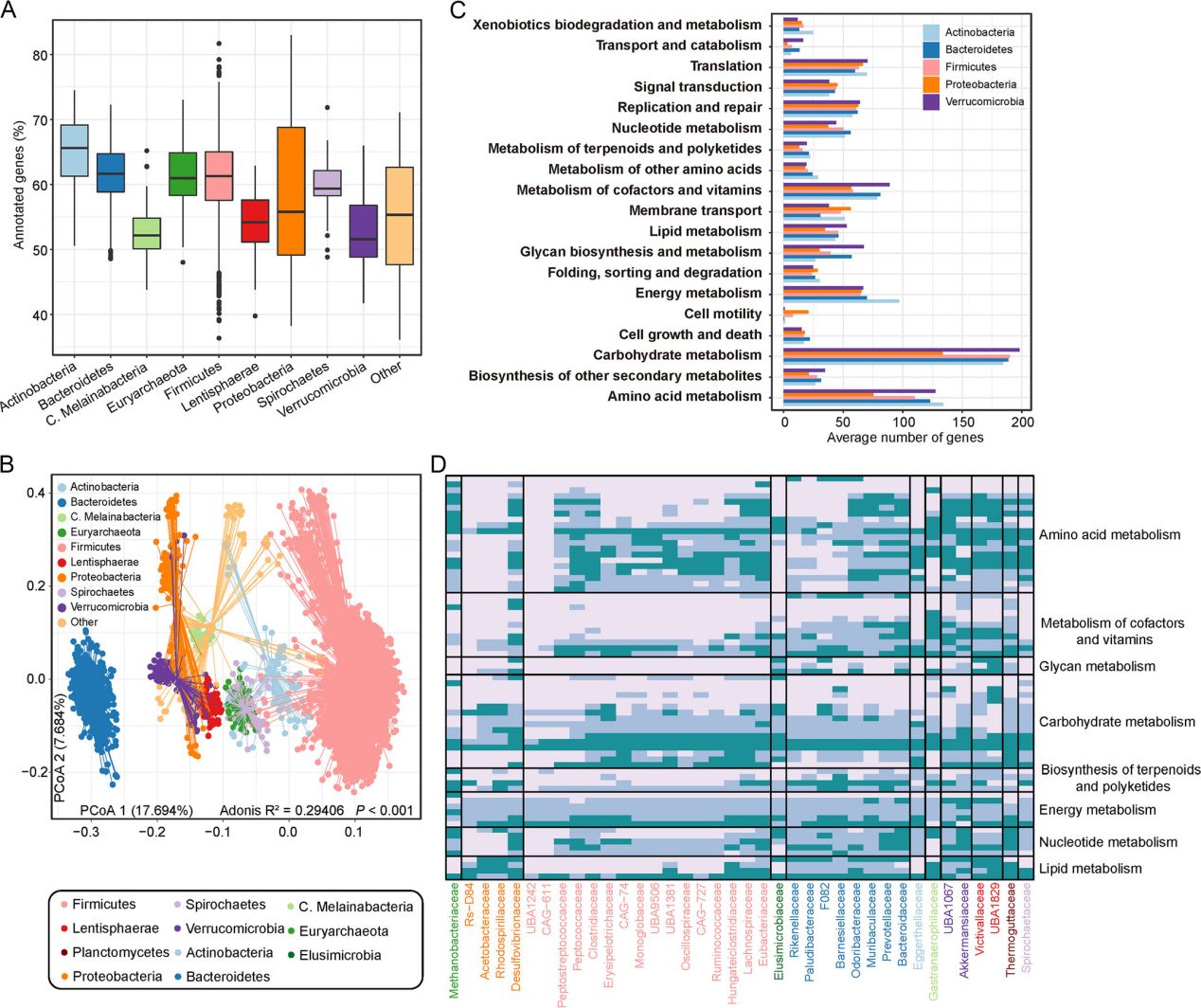
图3. Caprinae肠道MAG的KEGG图谱
4. 碳水化合物活性酶(CAZymes)的鉴定和描述
从Caprinae肠道MAGs中共获得960735个CAZyme编码基因。在GTs中,GT2和GT4亚科是两个主要成员,涉及的基因数量远远大于其他CAZyme。系统进化树显示,编码CAZyme的基因出现在主要家族中(图4)。厚壁菌门和拟杆菌共同贡献了数据集中的最大数量的CAZyme蛋白。
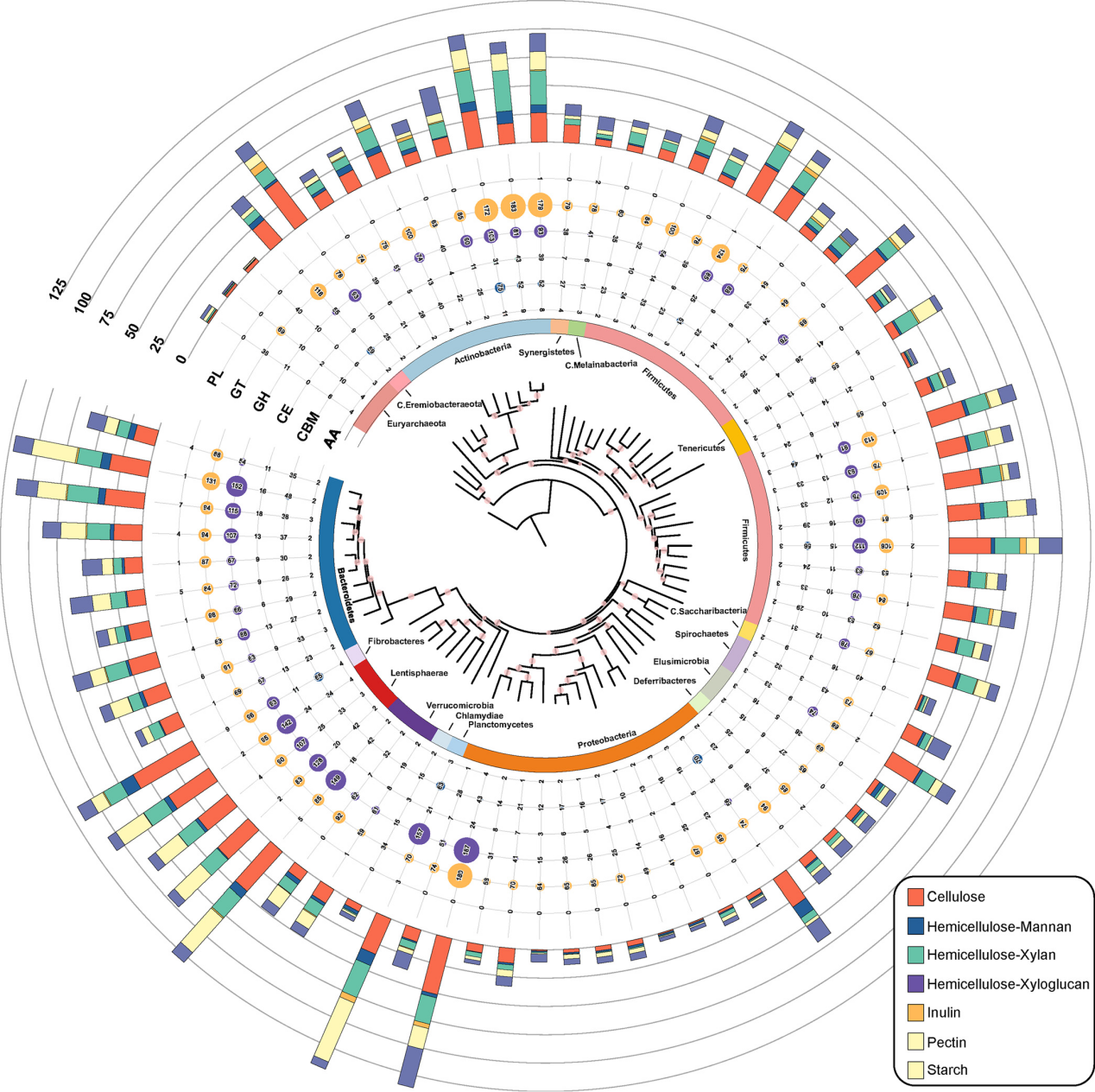
图4. Caprinae肠道MAGs的CAZyme和BGC概述
5. 微生物组成和功能的地理环境适应
PCoA分析表明,来自不同省份的样本的肠道微生物群特征存在显著差异(图5A)。多重方差分析表明,地理因素是肠道菌群变化的主要原因。不同地区样品的多样性无显著差异(图5B)。来自西藏的样品具有更多的反刍球菌科细菌,更少的Prevotelleaceae和Rikenellaceae(图5C)。来自六个区域的样品中共有260种通用物种(图5D),表明在这些Caprinae动物中存在共同的核心肠道菌群。与多样性分析一致,特有物种主要出现在西藏和广西省(图5D)。
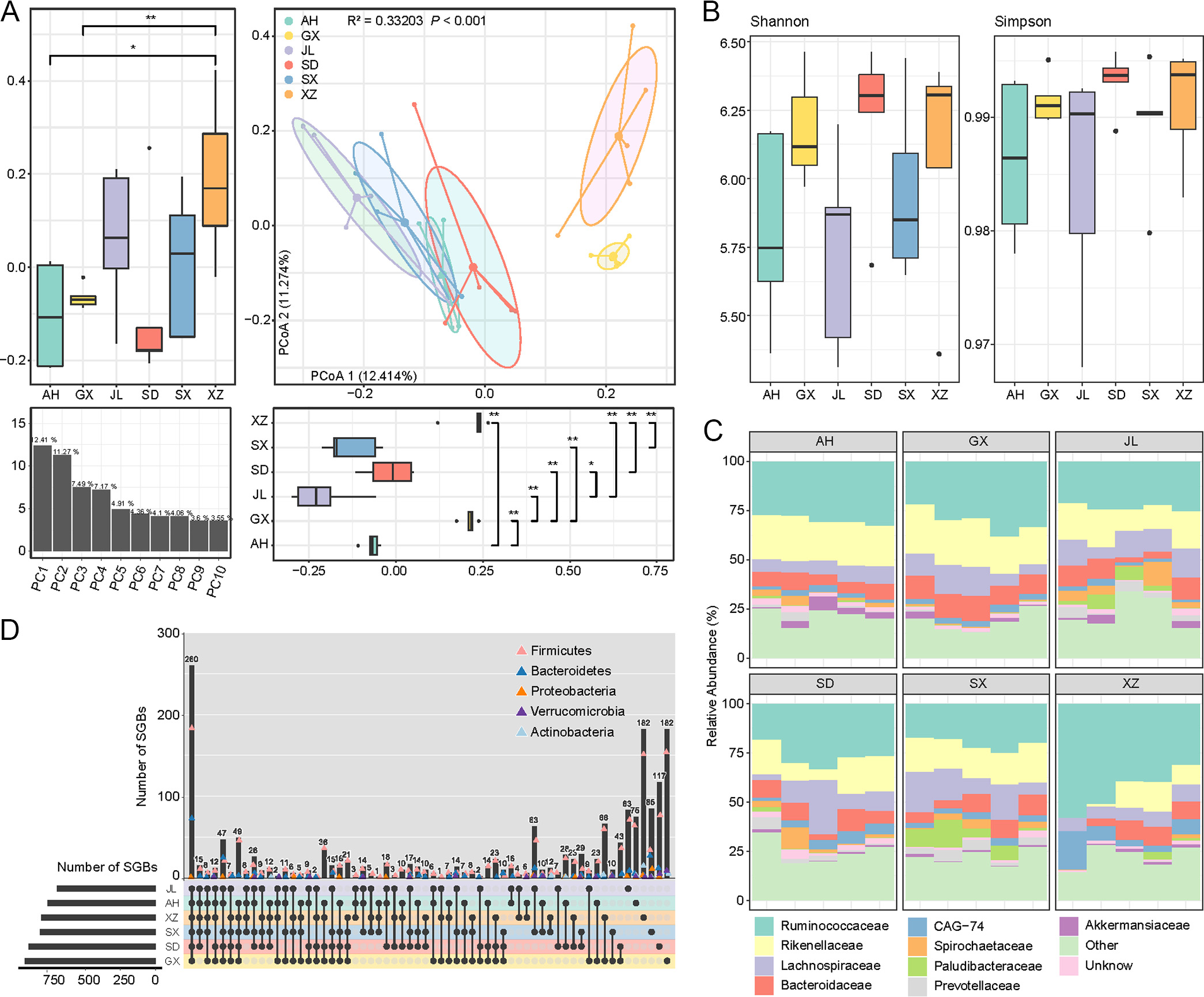
图5. 不同地理区域Caprinae肠道微生物组的比较
进一步讨论藏区的肠道微生物群特征,将所有藏区样本与非藏区样本进行了比较。共有186个高丰度SGBs在藏区样本中被观察到,81个在非藏区样本中为高丰度。藏区动物富含厚壁菌门、放线菌门和广古菌门,拟杆菌门相对较少(图6A)。同样的微生物群模式也存在于牦牛和山羊的肠道中,这可能与适应极端高原环境的能力有关。此外,比较了藏区和非藏区肠道菌群的功能特征。西藏动物具有更多的代谢相关模块,尤其是核苷酸代谢和氨基酸代谢。此外,藏区动物体内的细菌参与钴胺素、胆固醇、脂肪酸、麦角钙化醇等的合成比非藏区动物多。M00356和M00567被发现在西藏样本中富集,这些模块是产甲烷的关键(图6B)。编码K00320和K14081的基因在西藏样本中显著增加(图6C)。
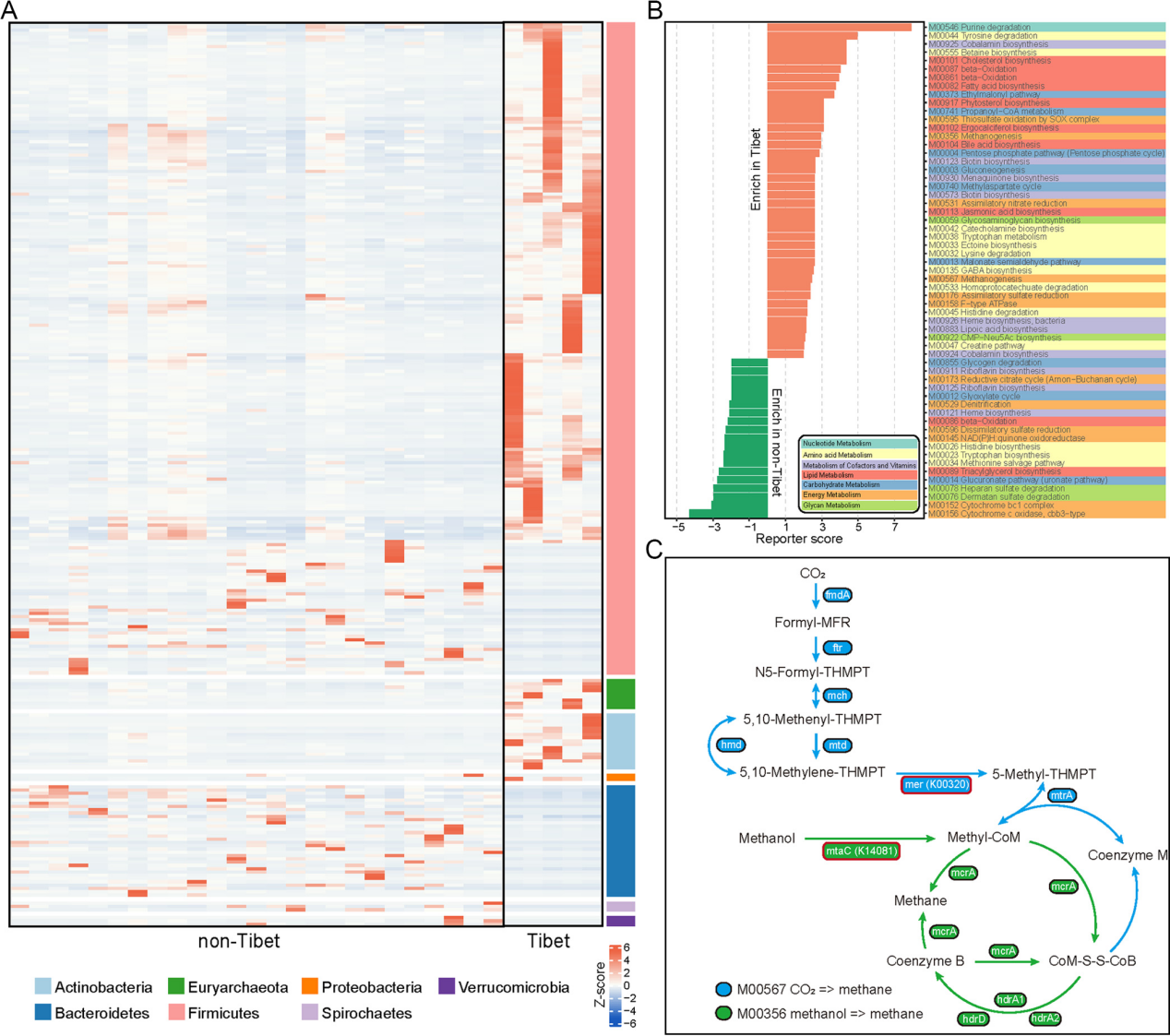
图6. 藏区和非藏区山羊肠道菌群组成和功能比较
全文总结
了解Caprinae的肠道菌群结构可以帮助设计适当的干预措施,以提高饲料转化率和个体健康。在未来的工作中,许多分离和培养的物种将成为研究肠道菌群功能的基础。肠道菌群的深层宏基因组测序和物种组装对于改善培养条件和调控肠道菌群至关重要。
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



