NP解读|单分子靶向可及性和甲基化测序揭示拟南芥着丝粒、端粒和rDNA的表观特征

英文标题:Single-molecule targeted accessibility and methylation sequencing of centromeres, telomeres and rDNAs in Arabidopsis
发表期刊:nature plants
发表时间:2023.8.20(IF:18)
通讯作者:南方科技大学生命科学学院生物系翟继先团队和中科院遗传发育所曹晓风课题组青年研究员邓娴
研究背景
目前ATAC-seq(转座酶染色质可及性测序)和WGBS(全基因组亚硫酸盐测序)已成为分析基因组的染色质可及性和甲基化的有效方法,但由于二代测序读长短,所以很难对着丝粒、端粒和核糖体DNA等高度重复区域(HRR)的甲基化和可及性进行准确测定。本研究基于ONT长读长靶向测序和甲基转移酶(EcoGII)相结合的策略,开发了植物中的单分子靶向染色质可及性和DNA甲基化测序(Single-molecule Targeted Accessibility and Methylation Sequencing, STAM-seq),并对野生型和DNA甲基化突变体拟南芥的HRR区域的表观遗传进行了表征。
材料方法
本研究对野生型(WT)和DNA甲基化突变体(met1, ddcc)拟南芥进行了细胞核分离,并利用6mA 甲基转移酶 EcoGII 对染色质可及性区域进行标记,进而采用ONT靶向测序技术进行测序分析。
研究结果
1. HRR区域的染色质可及性和DNA甲基化的靶向检测
通过运用ONT靶向测序技术,我们对HRR区域及其±100 kb范围内的序列进行了检测,并将此结果与非靶向测序数据进行了对比。结果显示,采用靶向测序方法后,HRR区域的测序覆盖率显著提升,实现了对该区域高达4.8倍的富集。将STAM-seq的数据与ATAC-seq和WGBS数据进行比较分析,结果显示STAM-seq检测到的可及性信号和DNA甲基化位点与ATAC-seq的可及性信号和WGBS的DNA甲基化位点显示出良好的一致性。
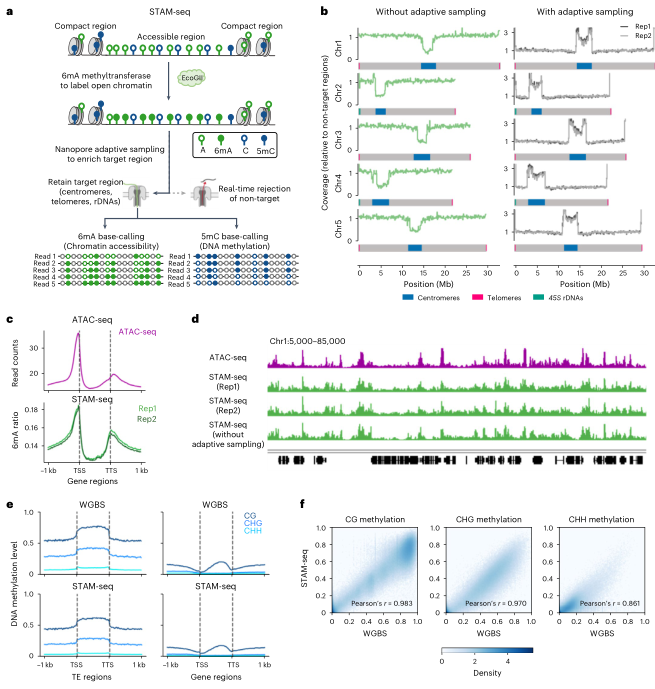
图1 STAM-seq分析HRR区域的染色质可及性和DNA甲基化
2. 着丝粒的链特异性表观遗传特征
在拟南芥中,着丝粒主要由CEN180卫星重复序列组成,本研究分别分析了着丝粒的正向链和反向链的表观遗传状态,发现正向链的可及性较高,CG和CHH甲基化水平较低。这与之前的研究一致,着丝粒中的链特异性甲基化可能对着丝粒形成、姐妹染色单体内聚或纺锤体附着具有潜在影响。
拟南芥CEN180卫星重复序列通常以头尾相连的方式串联形成高阶重复序列(HORs),多项研究表明,ATHILA逆转录转座子的入侵会导致HOR断裂。本研究发现,与相邻的CEN180重复序列相比,ATHILA区域的可及性降低,ATHILA中的CHG甲基化水平较高,表明ATHILA和CEN180重复序列的表观遗传景观不一致,这很可能是抑制ATHILA移动的原因。同时还观察到整个CEN180重复序列中的周期性分布,其特点是重复序列的中心区域与边缘相比具有更高的可及性和更低的 DNA 甲基化。
为了研究着丝粒区域DNA甲基化与染色质可及性之间的相互作用,本研究分析了野生型和DNA甲基化突变体拟南芥的着丝粒区域的甲基化和可及性,发现突变体的甲基化水平降低,染色质可及性增加,表明DNA甲基化有可能调节着丝粒染色质区域的染色质结构。
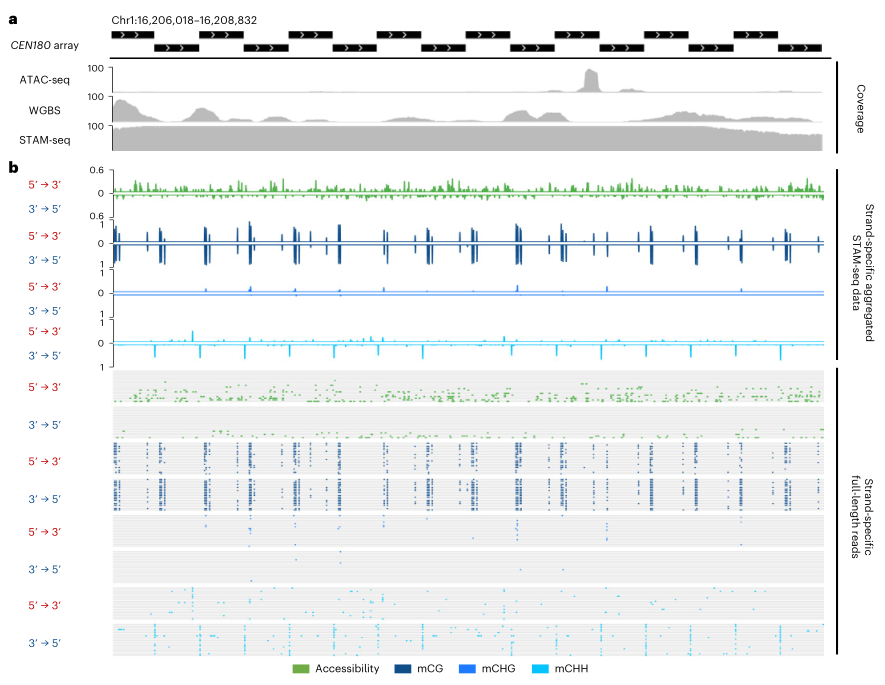
图2 着丝粒区域表观遗传模式的链特异性分析
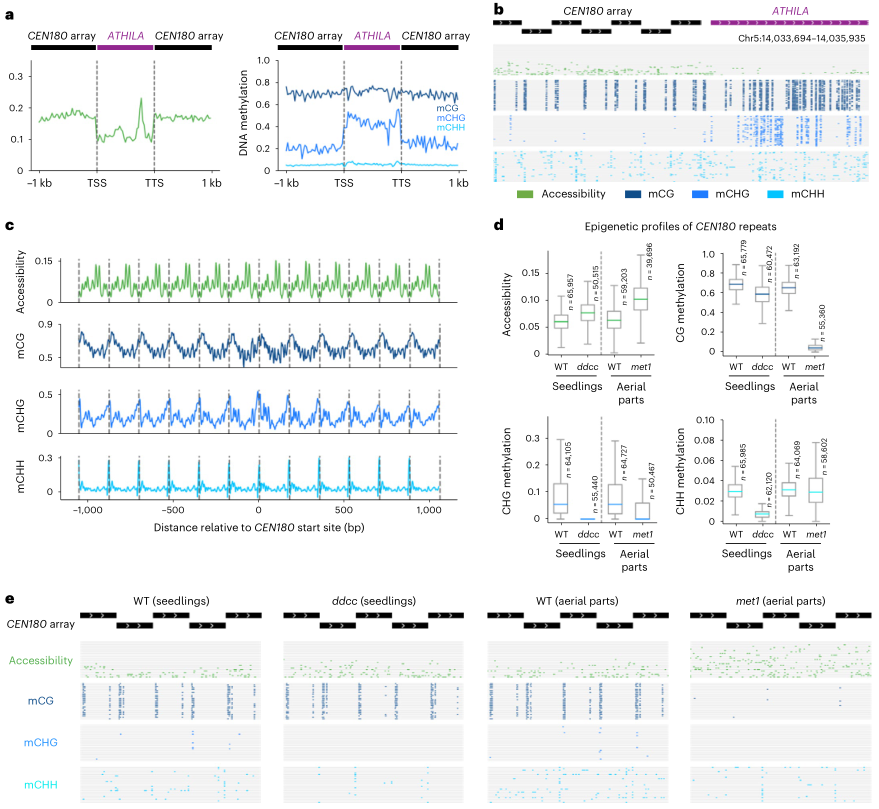
图3 着丝粒区域染色质可及性和DNA甲基化分析
3. 45S rDNA簇的表观遗传特征
根据外部转录间隔区,本研究将rDNA分为了3个主要类别(VAR1、VAR2 和 VAR3)和一个低拷贝数类别(VAR4),本研究发现同一类的rDNA往往聚集在一起,通常处于沉默状态的VAR1被大量甲基化。然而,转录活跃的VAR2和VAR3显示出较高的可及性和较低的DNA甲基化。同时还观察到同一类rDNA也显示出不同的表观遗传状态,可分为低甲基化和高甲基化两组,DNA甲基化低的组,染色质可及性高。对DNA甲基化突变体的进一步分析表明,DNA甲基化减少伴随着45S rDNA上染色质可及性的增加。总体而言,这些发现显示了DNA甲基化和染色质可及性之间呈负相关,为深入了解表观遗传调控的动态及其对rRNA表达的可能影响提供了见解。
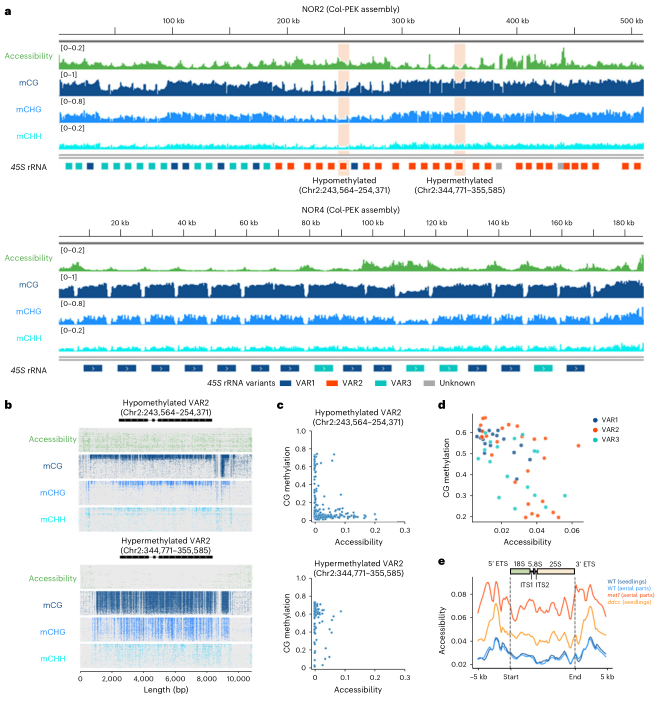
图4 STAM-seq揭示了rRNA基因的不同表观遗传特征
4. 端粒链上的非对称表观遗传特征
端粒区域由端粒序列自身及其相邻的亚端粒结构组成,位于染色体的末端,在拟南芥中,端粒由n个(CCCTAAA)序列串联重复组成。本研究分析了端粒和相邻的1kb的亚端粒区域,发现端粒的可及性和CHH甲基化水平都较低,亚端粒区域显示出较高的DNA甲基化特征。
链特异性分析表明,端粒reads在长度上呈现链不对称,G-rich链比C-rich链长,G-rich链的DNA甲基化水平高于C-rich链,这一现象可能与端粒DNA的半保留复制有关。同样在DNA甲基化突变体中观察到端粒区域的甲基化减少和可及性增加。
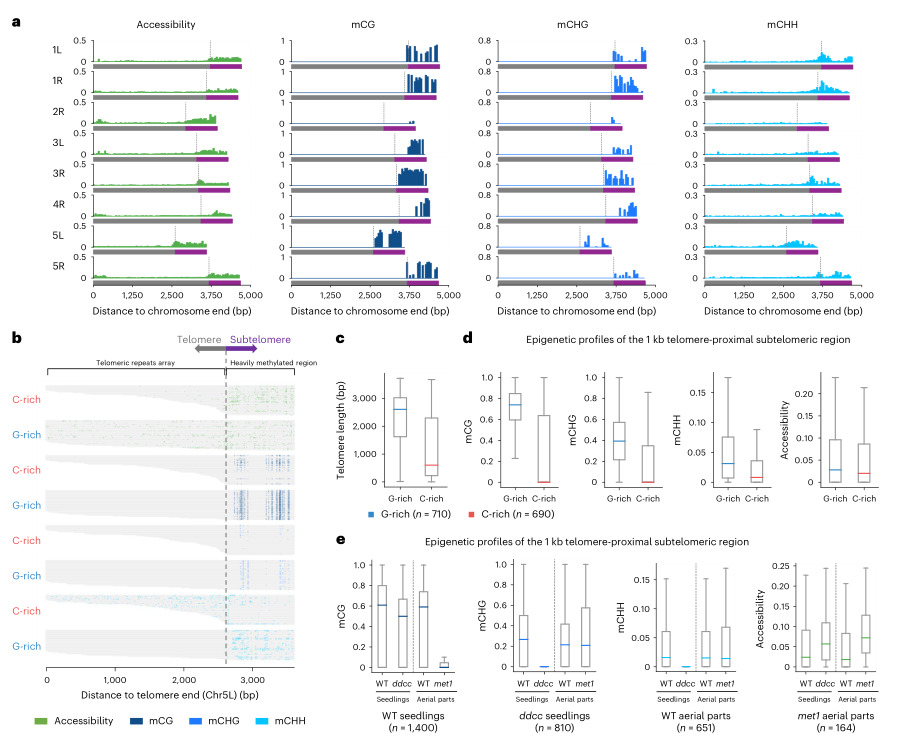
图5 端粒区域的染色质可及性和DNA甲基化
研究结论
本研究开发了单分子靶向可及性和甲基化测序(STAM-seq),用于研究野生型和DNA甲基化突变体拟南芥中的HRR区域,发现CEN180重复序列在其正向链上表现出较高的染色质可及性和较低的DNA甲基化水平,单个rDNA单位的DNA甲基化与可及性呈负相关,端粒的可及性和CHH甲基化水平均低于相邻的端粒亚区。STAM-seq提出了一种专为植物研究量身定制的方法,重点关注植物基因组,并有效研究HRR中的表观遗传特征。
参考文献:
Weipeng Mo, et al. Single-molecule targeted accessibility and methylation sequencing of centromeres, telomeres and rDNAs in Arabidopsis. Nature Plants. 2023
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



