T2T基因组系列(二):T2T基因组文献分享
上期为大家介绍了T2T genome(基因组4.0)掀起了后基因组时代的研究浪潮,并简要介绍了T2T基因组的研究策略和后续研究方向(详见:)。
简短的介绍使广大学者对T2T基因组研究领域产生兴趣,应读者的建议,T2T基因组系列第二期将为大家详细盘点目前已发表的T2T基因组高分文章,为各位研究学者提供高分T2T基因组研究思路。
文末含有本期福利(T2T基因组科研干货)
1.Nature(IF=49.962)——Telomere-to-telomere assembly of a complete human X chromosome.
论文题目:组装人类完整的端粒到端粒到X染色体
发表时间:2020.07
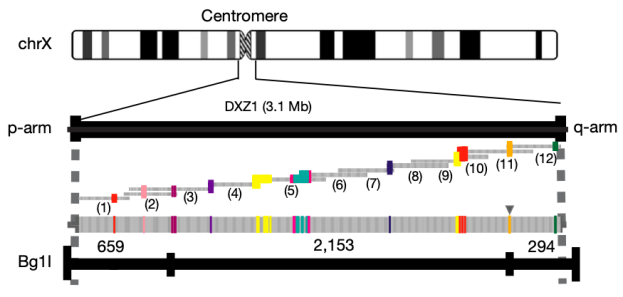
主要研究内容:使用ONT Ultra-long和PacBio测序技术,组装人类“CHM13”基因组,从中选择组装连续性较高的X染色体进行进一步研究。填补了X染色体组装中的29个gaps,共计1,147,861bp此前组装模糊的碱基。重组装了3.1Mb的着丝粒区域,这些区域包括假染色体区域,以及与癌症相关的 CT-X 和 GAGE 区域,这些区域为解析人类基因组相关疾病和癌症是至关重要的。此外,利用ONT测序数据,首次、完整绘制出复杂的串联重复序列和微卫星序列,包括它们的甲基化图谱。
文章链接://www.nature.com/articles/s41586-020-2547-7
参考文献:Miga KH, et al. Telomere-to-telomere assembly of a complete human X chromosome. Nature. 2020.
2.Nature(IF=49.962)——The structure, function, and evolution of a complete human chromosome 8.
论文题目:完整的人类8号染色体的结构、功能和进化
发表时间:2021.04
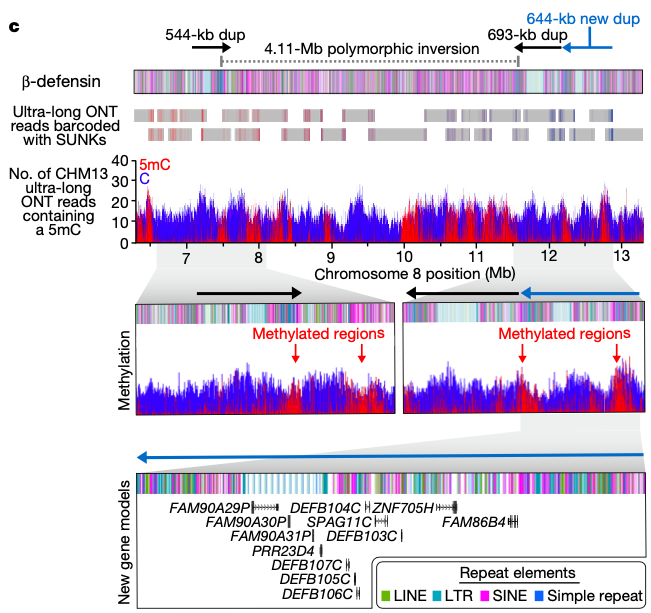
主要研究内容:使用ONT ultra-long和PacBio HiFi技术解决人类8号染色体的缺口,该组装解决了5个长期存在的gaps,包括2.08Mb的着丝粒α卫星序列,644kb的β-defensin多态性序列,863kb的8q21.2 VNTR序列以及长臂和短臂末端的端粒区。此外,该研究完成了黑猩猩、猩猩和猕猴的8号染色体高质量同源着丝粒的组装草图,重建了其进化历史,通过比较分析和系统发育分析,发现黑猩猩的第8号染色体的着丝粒组织结构与人类的最为相似。这些分析提供了灵长类同源染色体着丝粒的完整比较序列分析,为未来研究基因组中这些区域的遗传变异和进化提供了框架。
文章链接://www.nature.com/articles/s41586-021-03420-7
参考文献:Logsdon GA, et al. The structure, function and evolution of a complete human chromosome 8. Nature. 2021.
3.bioRxiv——The complete sequence of a human genome.
论文题目:人类完整基因组序列(首个人类基因组完成图——T2T-CHM13)
发表时间:2021.05
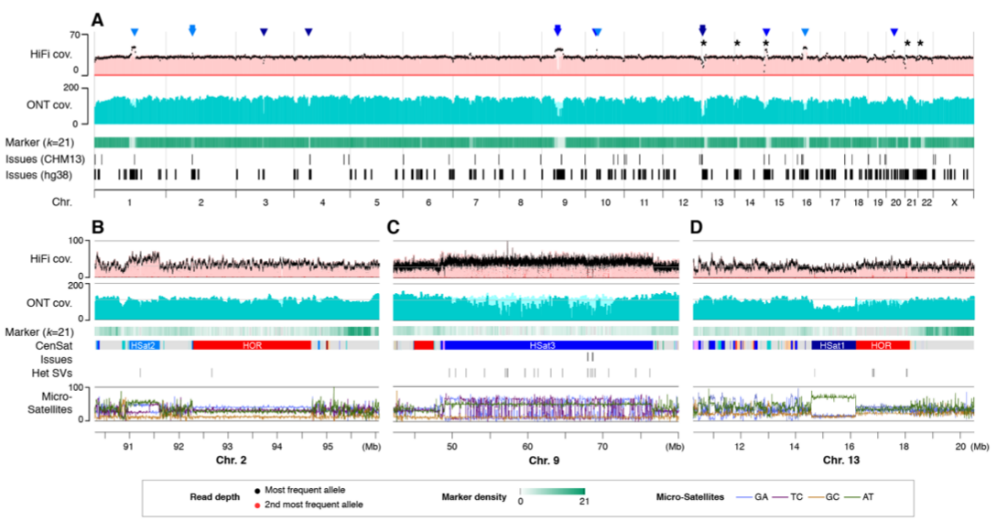
主要研究内容:自2001年首次公布人类基因组初稿以来,人类基因组被不断的更新和填补,但仍有复杂区域未被完成或其他错误。该研究是自首次发布人类基因组以来的最大改进,新的T2T-CHM13包括所有22个常染色体和X染色体的无Gap组装,纠正了许多错误,并引入了近2亿bp的新序列,新完成的区域包括所有的着丝粒序列和5个染色体短臂的端粒序列,首次将这些基因组的复杂区域用于变异和功能研究。
文章链接://www.biorxiv.org/content/10.1101/2021.05.26.445798v1
参考文献:Nurk S, et al. The complete sequence of a human genome. BioRxiv. 2021.
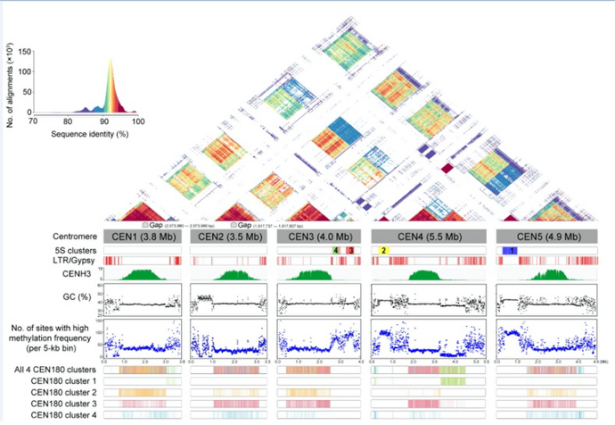
4. Science(IF=47.728)——The genetic and epigenetic landscape of the Arabidopsis centromeres
论文题目:拟南芥着丝粒的遗传和表观遗传
发表时间:2021.11
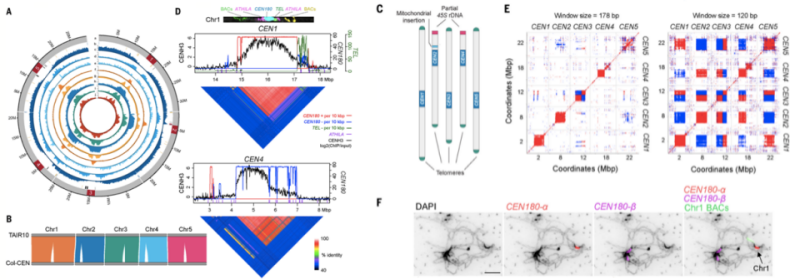
主要研究内容:该研究利用ONT和HiFi测序技术,重新组装了拟南芥Col-0的基因组,命名为Col-CEN v1.2,相比TAIR10基因组,Col-CEN中五条染色体的着丝粒序列被完整的组装。利用拟南芥Col-CEN基因组,研究者发现同一染色体着丝粒上的CEN180序列存在均质化的趋势,而ATHILA逆转录转座子的插入则破坏了CEN180序列的遗传学和表观遗传学结构,促进CEN180序列的多样化,均质化和多样化共同推动了拟南芥着丝粒的进化。
文章链接://www.science.org/doi/10.1126/science.abi7489
参考文献:Naish M, et al. The genetic and epigenetic landscape of the Arabidopsis centromeres. Science. 2021.
5. Genomics, Proteomics & Bioinformatics(IF=7.691)——High-quality Arabidopsis thaliana Genome Assembly with Nanopore and Hi‐Fi Long Reads
论文题目:利用Nanopore和HiFi长读长组装高质量拟南芥基因组
发表时间:2021.08
主要研究内容:该研究使用Nanopore ultra-long、PacBio HiFi以及Hi-C数据进行拟南芥基因组组装,新组装引入了14.6Mb的新序列,Col-XJTU组装的5条染色体均具有较高的Q V值,高于TAIR10.1参考。后续研究了着丝粒的结构和表观遗传学,检测到4个CEN180单体簇,发现着丝粒特异性组蛋白对CEN180簇表现出强烈的偏好,这一高质量的基因组为了解植物着丝粒多态性及遗传和表观遗传特征提供非常有价值的参考。
文章链接://doi.org/10.1016/j.gpb.2021.08.003
参考文献:Wang B, et al. High-quality Arabidopsis thaliana Genome Assembly with Nanopore and HiFi Long Reads. Genomics Proteomics Bioinformatics. 2021.
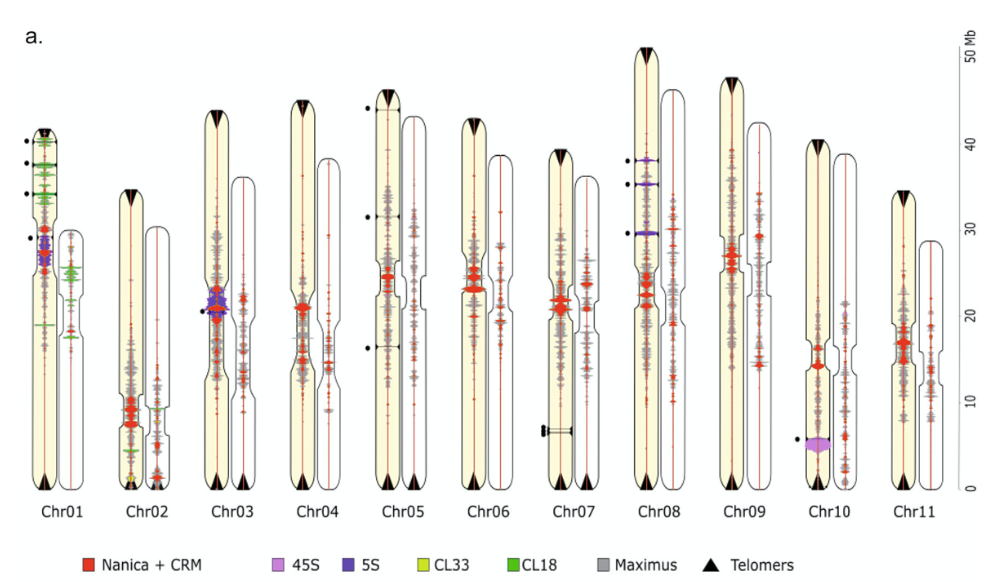
6. Communications Biology(IF=6.268)——Telomere-to-telomere gapless chromosomes of banana using nanopore sequencing
论文题目:香蕉基因组端粒到端粒组装
发表时间:2021.09
主要研究内容:该研究使用ONT ultra-long测序数据,完成了香蕉染色体水平基因组组装,在11条染色体中,有5条完成了端粒到端粒的组装,首次揭示了着丝粒和同源基因簇等复杂区域信息。基于香蕉T2T基因组,研究者新发现了1700个基因,这些新基因主要是串联重复基因,多以基因簇的形式存在于染色体重组区域,部分基因对香蕉的环境适应性和抗病性具有重要作用。此外,研究者还揭示了新组装的香蕉V4基因组与v1和v2版本间的共线性和结构变异,并对香蕉的A、B、S三套亚基因组的组装优化进行了初步探讨。该研究构建了香蕉基因组的近完成图,并解析了着丝粒和同源基因簇等复杂区域的组成。
文章链接://www.nature.com/articles/s42003-021-02559-3
参考文献:Belser C, et al. Telomere-to-telomere gapless chromosomes of banana using nanopore sequencing. Commun Biol. 2021.
AG代理基因以推动物种基因组研究进入新时代为己任,长期致力于基因组测序和组装技术的研发,实现了N50>100Kb超长序列的建库测序稳定产出并开发了相应组装流程。
本期科研干货:关注“AG代理基因”公众号,后台回复“AG代理T2T”便可获得所有T2T基因组高分文献PDF合集。
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



