有何稀奇?独家ONT-全长16S开启微生物多样性3.0时代
致力微生物多样性研究的你,还在为二代16S的种属水平注释率低而愁苦?还在为PB-全长16S的5千条ccs数据量不饱和而郁闷?
大可不必!!
ONT-全长16S,基于Nanopore进行长读长测序,直接得到16S V1-V9全长序列,不惧种属水平注释率,真实呈现样本菌群结构
ONT-全长16S,基于Nanopore的超高通量,5万条reads只需200+元,轻轻松松搞定饱和度曲线,完整反映样本菌群结构
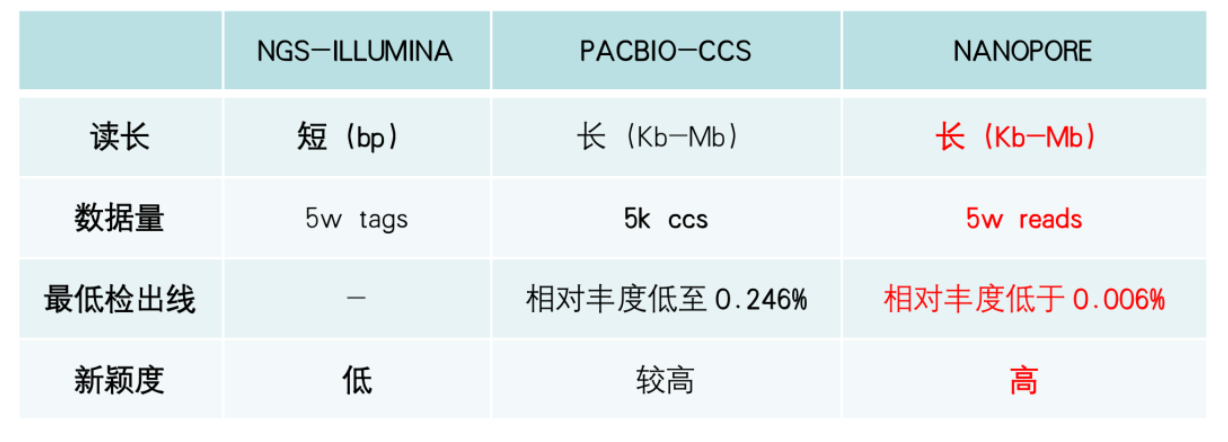
Tips:测16S V1-V9全长与测16S V3-V4等部分区域相比,差别多大?
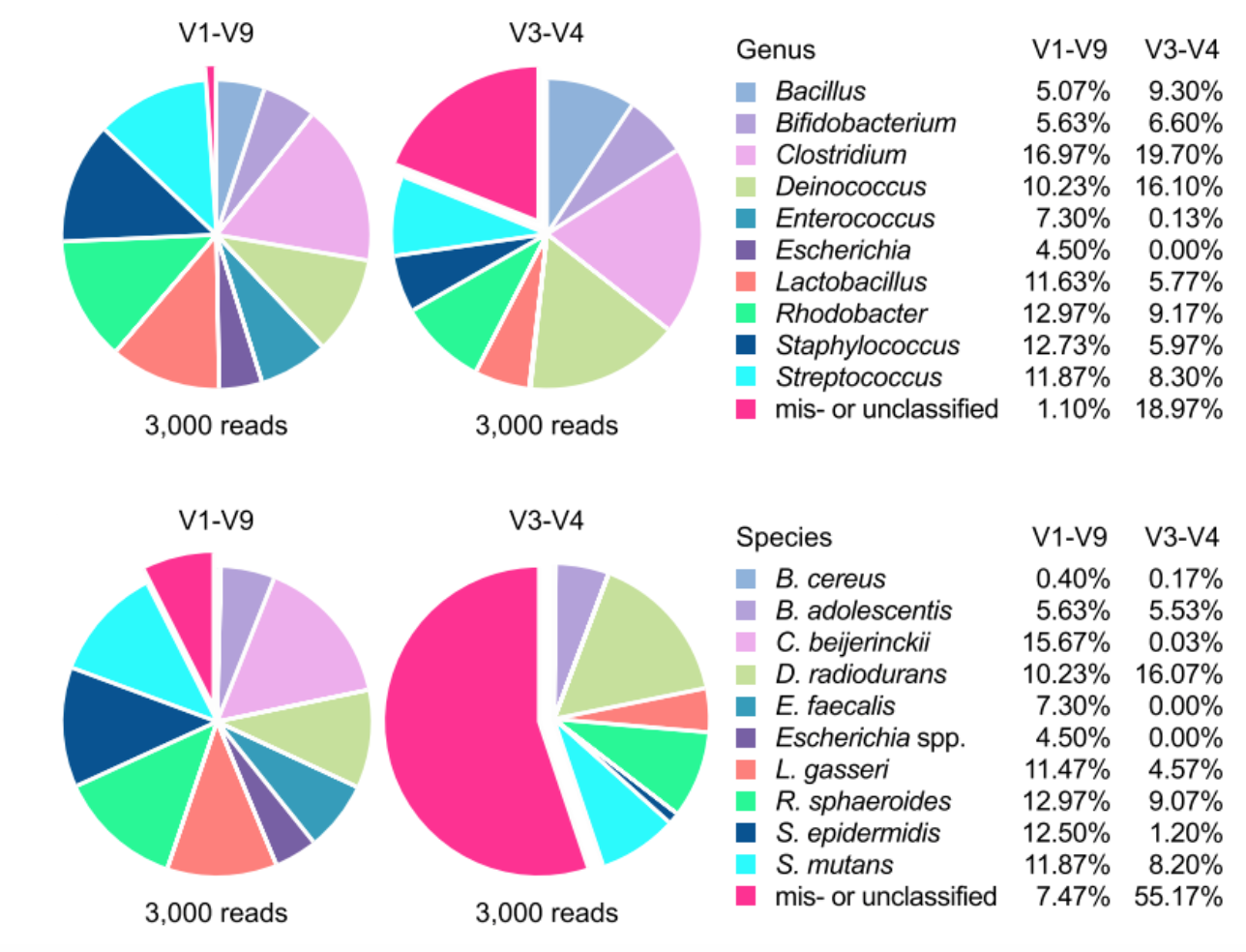
• 模拟菌群ONT-16S的结果中,大多数reads被分类到种属水平,且错误配对占比远低于二代16S。[1]
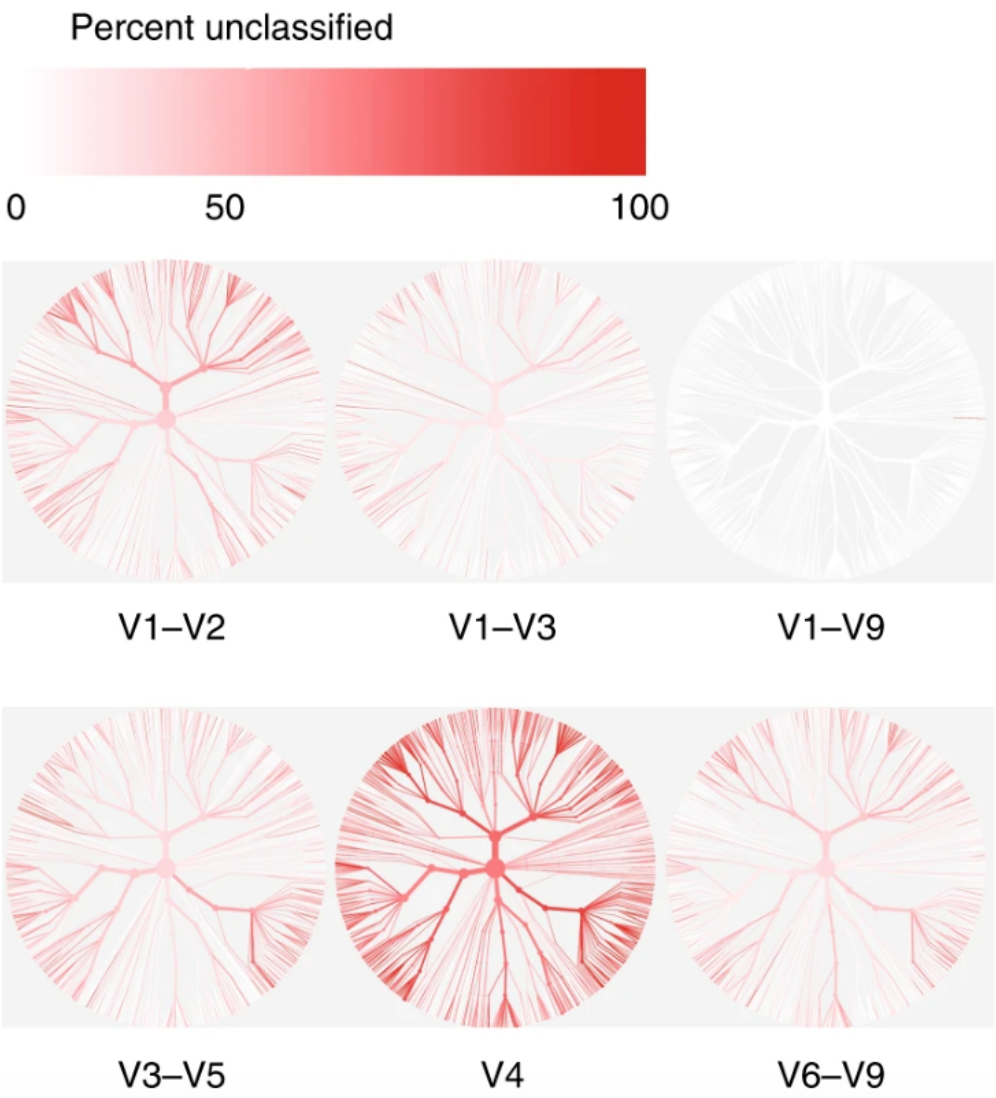
• 基于公共数据分析,分支的颜色越深说明种水平无法识别的序列越多,16S全长的注释率最高。[2]
[1] Matsuo, et al. Full-length 16S rRNA gene amplicon analysis of human gut microbiota using MinION nanopore sequencing confers species-level resolution[J]. BMC microbiology. 2021.
[2] Johnson J S, et al. Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis[J]. Nature Communications. 2019.
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



