文献解读 | Nanopore高质量基因组结合单细胞测序揭示了牦牛适应性进化的遗传机制
研究简介
牦牛是生活在世界平均海拔最高地区的、体型最大的反刍动物之一,在生理上能够适应寒冷和缺氧的环境。但是目前,牦牛适应极端条件的遗传和细胞基础仍然难以确定。家牦牛通常生活在海拔3000至5000米之间,而家牦牛的祖先野牦牛则栖息在海拔4000至6000米之间,均被认为是研究大型不如动物缺氧耐受性的优秀模型。以前对牦牛基因组的研究主要集中在使用短读长测序技术来发现单核苷酸变异,以揭示基因组多样性和历史种群动态。本研究使用长度长测序技术组装了两个染色体水平的基因组,分别用于野生牦牛和家养牦牛的研究,还使用单细胞转录组测序技术来揭示了一系列与牦牛肺独特结构发育相关的基因和途径。总之,这些这些数据为了解牦牛适应性进化背后的遗传和细胞机制提供了重要信息。
文章题目:Long read genome assemblies complemented by single cell RNA-sequencing
reveal genetic and cellular mechanisms underlying the adaptive evolution of yak
发表期刊:nature communications(IF=17.694)
发表时间:2022.09.06
主要研究结果
1.野生牦牛和家养牦牛的染色体水平基因组
使用Nanopore测序技术,野生牦牛和家养牦牛分别获得了157.17Gb和191.17Gb的数据, contig N50分别达到39.41Mb和46.79Mb;同时利用illumina短读数据(野生牦牛-114.38X;家养牦牛57.11X)来纠错;另外,使用Hi-C数据(野生牦牛-109.26X;家养牦牛143.56X)将contig锚定到染色体上。最后,成功组装出大小分别为2.63Gb和2.61Gb的野生和家养牦牛基因组,且二者都被定位到了30条染色体上。接下来,作者还评估了这两个基因组的序列一致性和完整性。数据显示,野生牦牛的比对率和覆盖率分别为99.35%和99.19%,而家养牦牛的比对率和覆盖率分别为99.18%和99.59%;BUSCO评估显示,野生牦牛和家养牦牛分别含有93.3%和93.2%的完整单拷贝基因,故表明其序列一致性和完整性较高。
2.野生牦牛和家养牦牛重复序列和基因的注释
对牦牛的重复序列和基因进行注释发现,野生牦牛和家养牦牛分别含有45.81%和45.67%的重复序列。选择了6种相关反刍动物物种进行基因同源性预测,结果在野生牦牛和家养牦牛基因组中分别预测到了22931和23143个基因,基于文库中的基因结构预测蛋白序列推断,估计在野生牦牛和家养牦牛中分别有95.4%和95.3%的基因具有功能。使用已知文库作为参考,还筛选了野生和家养牦牛基因组中的ncRNAs。
3.通过比较牦牛和黄牛基因组鉴定结构变异
长度长测序已被证明对SV的识别非常有效,本研究检测了3头黄牛、19头家养牦牛和7头野生牦牛的SVs,并与黄牛基因组进行比较,共检测到74279个缺失、79467个插入、3464个重复和867个反转。对所有SV注释发现,50.58%的SV位于内含子区,其他的SV在启动子、基因间、外显子、UTR或基因的上下游均有分布。为了研究高原适应,研究人员计算了黄牛和牦牛之间所有SV的FST,并选择FST值最高的0.5%的SV作为候选位点。有趣的是,作者发现这些候选SV的FST值高达1。最终,该研究注释了6733个携带高FST SV的基因和897个携带位于外显子和启动子区域的特殊SV基因(图1)。
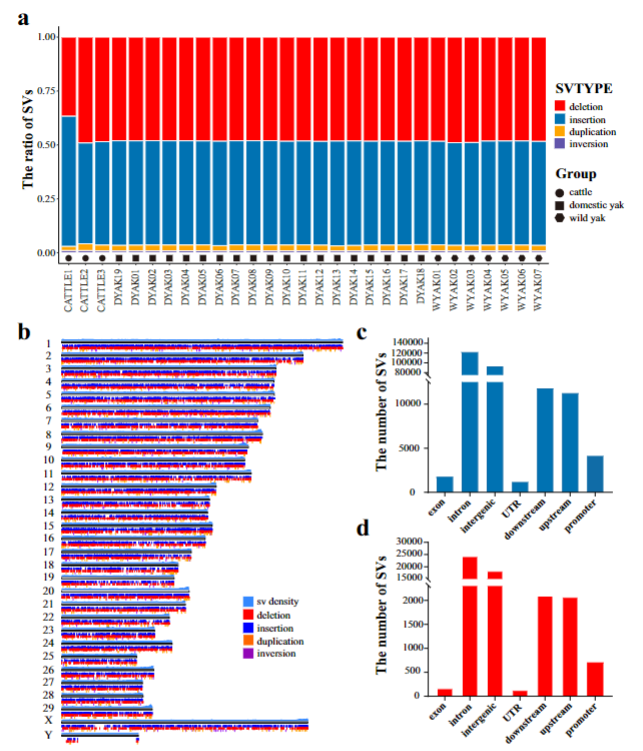
图1 牦牛和黄牛基因组结构变异(SV)的检测和表征
4.整合转录组和染色质可及性数据揭示潜在功能性SVs
为了探讨SVs在影响基因表达中的潜在作用,作者对来自48个样本的转录组数据进行分析,发现127个在外显子和启动子区域携带高FST SV的基因在黄牛和家养牦牛的肺中差异表达;对携带高FST SV的启动子区的peak进行motif富集分析,鉴定到了对缺氧反应重要的5种转录因子。总之,这些数据表明缺氧相关基因启动子中的SVs在基因表达调控中具有潜在功能(图2)。
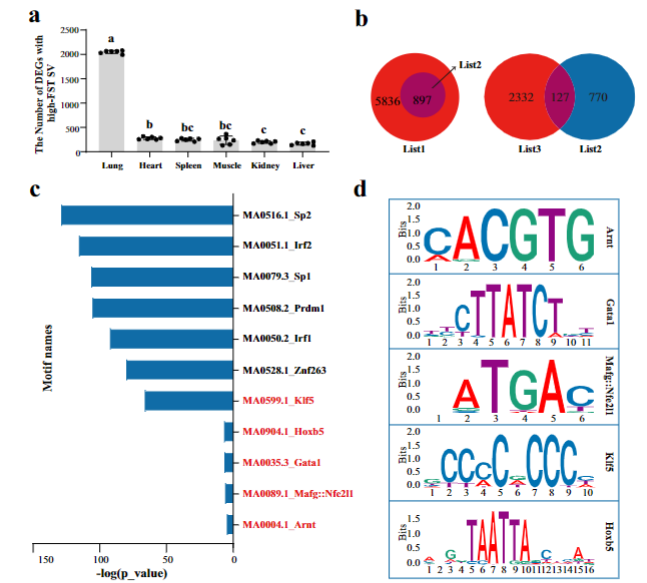
图2 牦牛高原适应候选基因的筛选
5.家养牦牛和黄牛肺组织的单细胞RNA-seq分析
尽管肺是介导对缺氧的生理适应的最重要器官之一,但尚未在高海拔动物的肺中研究单细胞水平上细胞群和基因表达谱的多样性。为此,本文消化了家养牦牛(n=5)和黄牛肺组织(n=5),并获得了用于RNA序列分析的单细胞。在两个文库构建之后,总共收获了31579个家养牦牛细胞和27951个黄牛细胞并进行了测序和单细胞图谱构建,将单细胞转录组数据分别细分为21和20个细胞群。综合基因组学和转录组学分析,揭示了一种新的内皮细胞亚型,并揭示了一系列与牦牛肺独特结构发育相关的基因和途径(图3)。
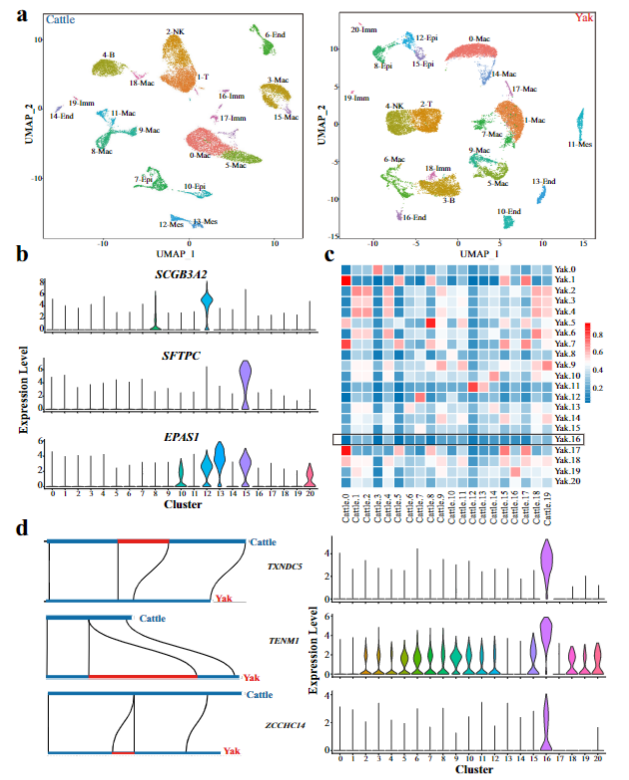
图3 单细胞RNA-seq在黄牛和家牦牛肺中发现了不同的细胞群
研究结论
总之,该研究提出了野生牦牛和家养牦牛的两个高质量染色体水平基因组,通过黄牛和牦牛的基因组比较来筛选携带高FST SV的候选基因,并进一步说明这些基因在牦牛适应中的潜在作用。还提供了牦牛中SV的全基因组目录和牛肺组织的单细胞转录组谱。这些高质量的基因组和单细胞RNA-seq数据为未来牛物种研究提供了重要来源。
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



