文献解读| Direct RNA测序揭示速生毛竹幼苗的表观遗传修饰
随着测序技术的发展,旨在探索竹子速生的组学研究已经越来越多。然而,当采用依赖PCR扩增的测序方法时,不能直接检测表观遗传修饰。牛津纳米孔技术公司开发的Direct RNA测序(DRS)方法可对单个RNA分子进行测序,得到包含修饰信息的转录本序列。因此,将DRS应用于毛竹幼苗可以进一步了解它们的速生机制。

文章题目:Comprehensive profiling of epigenetic modifications in fast-growing moso bamboo shoots
发表期刊:Plant Physiology
IF:8.005
发表时间:2022.11.23
研究背景:
毛竹幼苗的速生是由每个节间的快速伸长引起的。然而,其潜在的细胞过程和表观遗传机制在很大程度上仍未被发现。本研究作者采用了包括全基因组亚硫酸氢盐测序(BS-seq)和DRS在内的多组学方法,探究了毛竹速生过程中DNA/RNA甲基化和转录后调控。该研究将有助于揭示表观遗传修饰和转录后调控在毛竹速生中的作用,并有助于我们对植物速生过程进行全面理解。
材料与方法:
本研究以2m和4m高的毛竹幼苗为实验材料,取第18节间底部和中部组织,共四组样本进行BS-seq和DRS测序。
研究结果:
1.毛竹速生伴随着明显的形态变化
通过毛竹幼苗节间长度的测量结果,选择2m和4m幼苗的第18节间作为实验材料,并将底部和中部节间区域分别命名为S-2-B、S-2-M、S-4-B和S-4-M。石蜡切片的研究结果显示节间组织主要由薄壁细胞、纤维细胞、次生木质部和筛管组成。四种组织在细胞大小、细胞核数量和其他特征方面存在差异(图1C)。
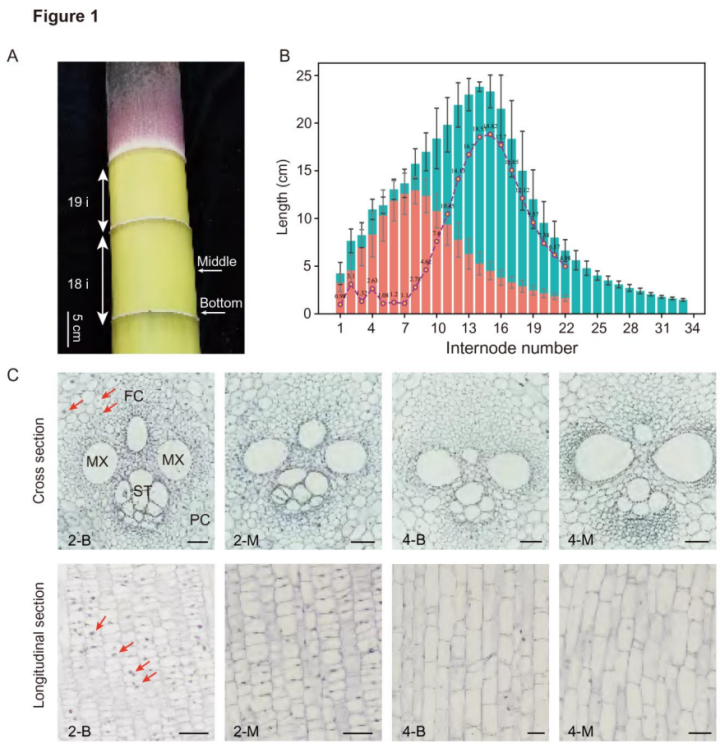
图1 毛竹伸长节间的生长模式及细胞形态
2.少量全长mRNA在毛竹幼苗速生过程中被转录
为了探究毛竹全长转录本的动态变化,进行DRS测序。DRS全长reads比例显示,2m幼苗的全长reads比例大于4m幼苗的(图2B)。这表明在毛竹幼苗中,mRNA降解可能随着高度的增加而增加。
3.DRS揭示了复杂的可变剪切事件
通过DRS检测到了多种可变剪切(AS)事件,其中大多数为内含子保留(IR)事件(图2D)。而IR基因的表达水平平均值(TPM)相对于非IR基因更高(图2G)。此外,大多数转录本仅显示来源于一种类型的AS事件,而发生多个AS事件的基因中,最多的是IR和5′可变剪接位点(A5SS)/3′可变剪接位点(A3SS)组合(图2F)。
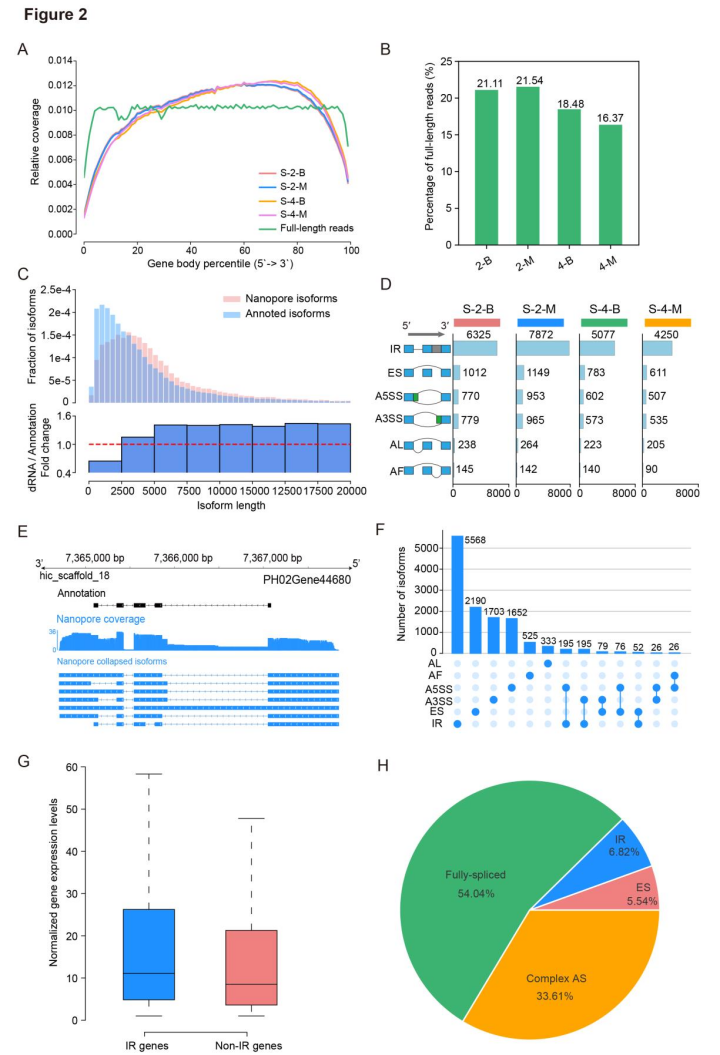
图2 全长转录本的特征和AS事件
4.3'UTR在毛竹幼苗速生期间全面延长
实验结果确定了每个基因3'UTR上所有Poly(A)位点的相对位置和丰度,并选择了两个丰度最高的多聚腺苷酸位点(PAS),根据其位置分别命名为近端PAS (pPAS)和远端PAS (dPAS)(图3A)。与S-2-B相比,S-4-B的转录本倾向于使用dPAS,这表明随着速生的进行,3'UTRs在节间底部区域延长(图3B)。GO富集分析表明,S-2-B和S-4-B组织之间发生PAS转换的基因主要与蛋白质翻译和转运相关(图3D)。
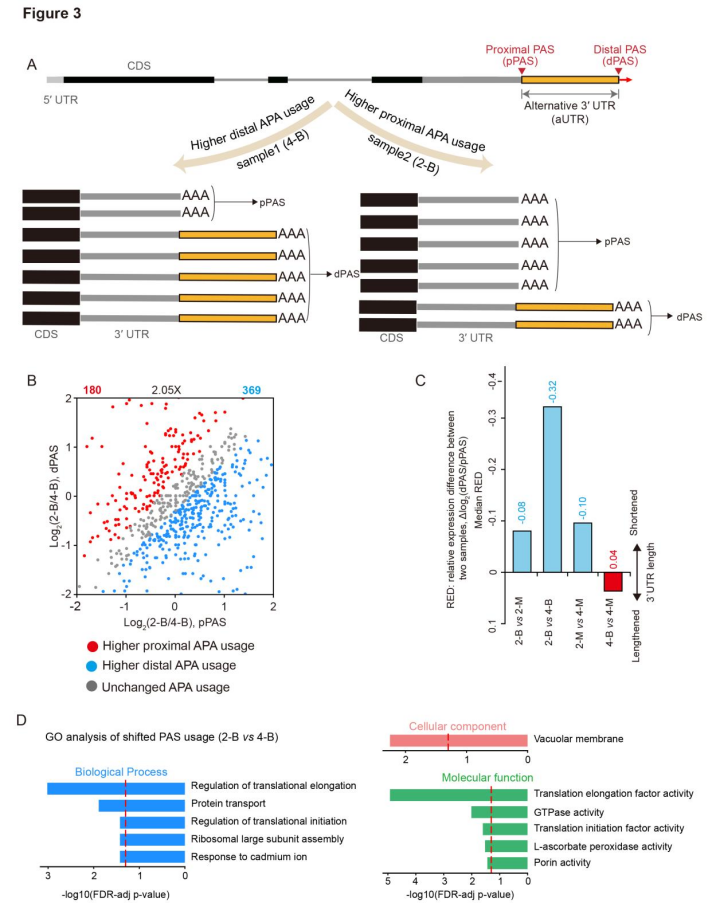
图3 毛竹速生期间poly(A)位点使用的动态变化
5.Poly(A)尾长在速生过程中的变化
DRS能够揭示Poly(A)尾长(PAL)在毛竹幼苗速生过程中的动态变化。通过比较样本PAL的中位值发现2m幼苗的PAL中位值比4m幼苗的长(图4A)。探讨PAL与转录本稳定性的关系,发现全长reads的中位PAL比非全长reads略长(图4B)。通过S-2-B vs S-2-M、S-2-B vs S-4-B、S-2-M vs S-2-M和S-4-B vs S-4-M的比较,鉴定出了具有较长和较短PALs的基因数量(图4C)。
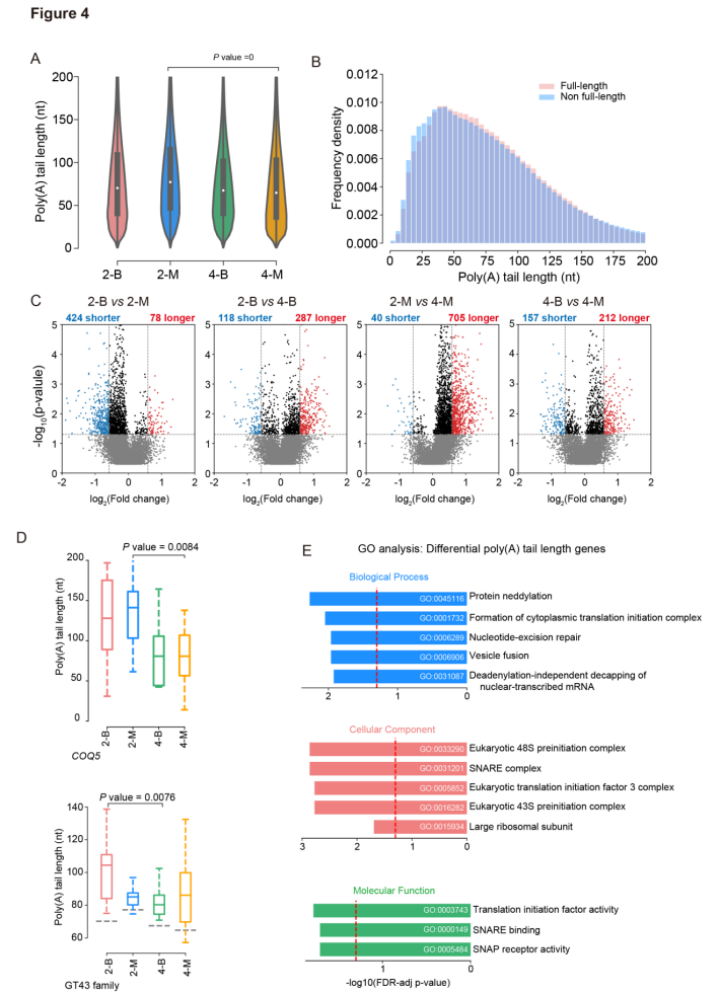
图4 毛竹在速生过程中Poly(A)尾长的分析
6.RNA的m6A修饰在速生过程中发生变化
本研究基于DRS数据分析了四种样本的m6A修饰的转录本数量并进行差异表达分析 (图5A)。不同组织的中位修饰率差异不大,但2m幼苗的m6A修饰率略高于4m幼苗(图5B),说明在毛竹的速生过程中,m6A修饰位点趋于逐渐去甲基化。观察m6A修饰位点在修饰的转录本中的分布,发现大多数m6A修饰存在于编码序列(CDS)内(图5C)。而转录本上m6A修饰位点的标准化分布表明,m6A在终止密码子和3'UTR附近大量富集(图5D)。另外,含有m6A修饰位点的外显子明显比未修饰的外显子更长(图5E),这与之前报道的m6A修饰通常发生在长外显子中是一致的。全长reads的m6A比率中位数高于非全长reads(图5F)。在S-2-B和S-4-M之间,具有不同APA使用情况的基因的m6A比例中位数高于无APA转移的基因(图5G)。

图5 基于纳米孔DRS的毛竹m6A修饰分析
7.转座元件和编码基因的DNA甲基化水平在毛竹幼苗速生过程中升高
为了了解幼苗速生过程中DNA甲基化的动态,进行了BS-seq。在转座子(TEs)中检测到高水平的DNA甲基化(图6A),而侧翼区域的甲基化水平较低。CG甲基化在蛋白质编码基因及其±2 kb侧翼区域呈钟形分布(图6B)。研究甲基化与基因表达水平之间的关系,发现非表达基因/低表达基因体的CHG和CHH甲基化水平高于高表达基因体(图6C)。另外,蛋白质编码基因的CG甲基化降低与较短的PALs相关,而CHG和CHH的甲基化水平没有显示出类似的模式(图6D)。
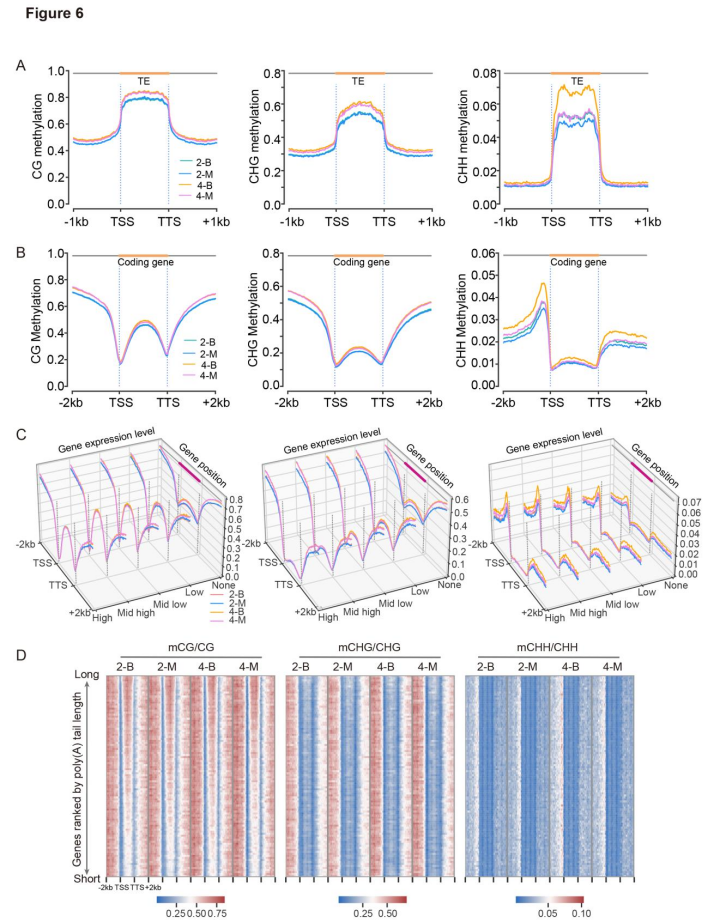
图6 基于BS-seq的毛竹DNA甲基化谱
8.DNA在速生过程中甲基化程度不同
为了研究不同高度幼苗甲基化特征的差异,鉴定了不同甲基化区域(DMRs)。在S-2-B vs S-2-M和S-4-B vs S-4-M比较中检测到大多数DMRs与甲基化损失相关(图7A),这表明无论幼苗高度如何,节间中间区域的DNA甲基化水平都低于底部。CHG DMRs是最丰富的DMRs类型,其次是CG DMRs和CHH DMRs(图7B)。少量基因同时携带三种类型的DMRs (图7C)。另外基因的CG DMRs密度高于上游或下游区域,CHG DMRs基因两侧的密度高于基因内部的密度,CHH DMRs表现出相反的模式(图7D)。进一步将转录区域分为内含子和外显子,发现内含子含有的DMRs远多于外显子(图7E-F)。
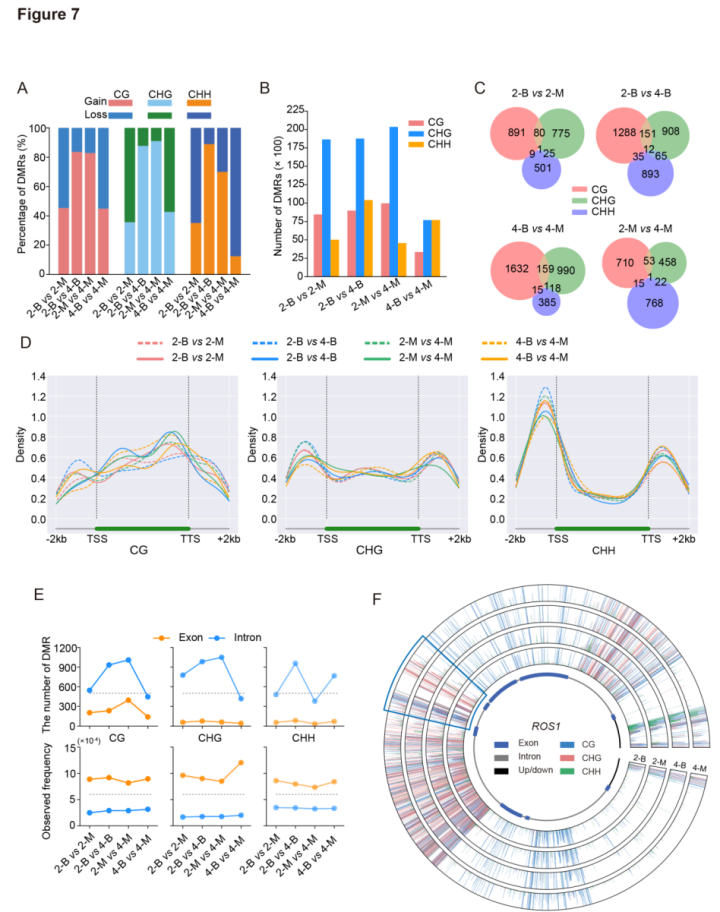
图7 差异DNA甲基化谱的全局分析
研究结论:
本研究基于Direct RNA测序和BS-seq,探究了毛竹速生过程中全长转录本、可变剪切、APA以及DNA/RNA甲基化的动态变化,揭示了表观遗传修饰和转录后调控在毛竹快速生长中的作用,有助于我们对植物速生过程进行全面理解。
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



