文献解读 | 长读宏基因组组装基因组改进了活性污泥中新型完整BGCs的识别
微生物产生各种各样的次生/特化代谢物(SMs),是抗生素的主要来源,并被用于一系列生物技术应用,然而其中大多数尚未被发现。生物合成基因簇(BGC)的跨度为几十kb到200 kb以上,精确的BGC检测需要完整的基因组组合,这在技术上很难从宏基因组中获得,直到有了新的长读测序(Oxford Nanopore)技术。本研究通过挖掘从长读数据中生成的高质量宏基因组组装基因组,确定了活性污泥的生物合成潜力。表明使用长读测序的高质量 MAGs组装对于探索微生物群落中未培养成员产生SMs的遗传潜力至关重要。

文章标题:长读MAGs改进了复杂微生物活性污泥生态系统中新型完整生物合成基因簇的识别
发表期刊:mSystems (IF=7.324)
发表时间:2022.10.30
研究策略
1. 数据收集
从早期研究中选择了1080个活性污泥细菌的高质量MAGs用于BGC挖掘;从GTDB-R202数据集中选择了粘菌(583个基因组)和硝化螺旋菌(328个基因组)的代表性基因组。
2. 基于GTDB的高质量 MAGs分类学鉴定
用基于数据库版本GTDB-R202的GTDB-tk v1.7.0工具包的classify_wf工作流分配了分类学特征。
3. 基因组挖掘分析在高质量 MAGs中检测BGCs
数据集中的所有高质量 MAGs都使用Prokka重新注释,使用Prodigal进行ORF检测。利用软件antiSMASH检测BGCs。
主要研究结果
1. 来自活性污泥的高质量MAGs具有丰富的生物合成潜力
MiDAS基因组数据库包括1080个细菌的高质量MAGs,包括30个门中的578个物种,大多数属于未培养和未鉴定的物种。antiSMASH检测到的4238个BGCs中,有3714个是完整的。最常见的预测产物是萜烯、rip -like SMs和芳基多烯(图1)。在所有门中都检测到BGCs,每个基因组的BGCs中位数为4个或更少(图1)。MAGs GCA_016714225.1属于黏菌科未知属,BGCs最多,共有23个,其中完整的有20个。此外,硝化螺旋体MAGs也表现出很高的生物合成潜力。该门包括亚硝酸盐氧化细菌(NOB)和完全氨氧化菌(comammox),它们对活性污泥的性能至关重要,但它们的生物合成潜力大部分仍未开发。
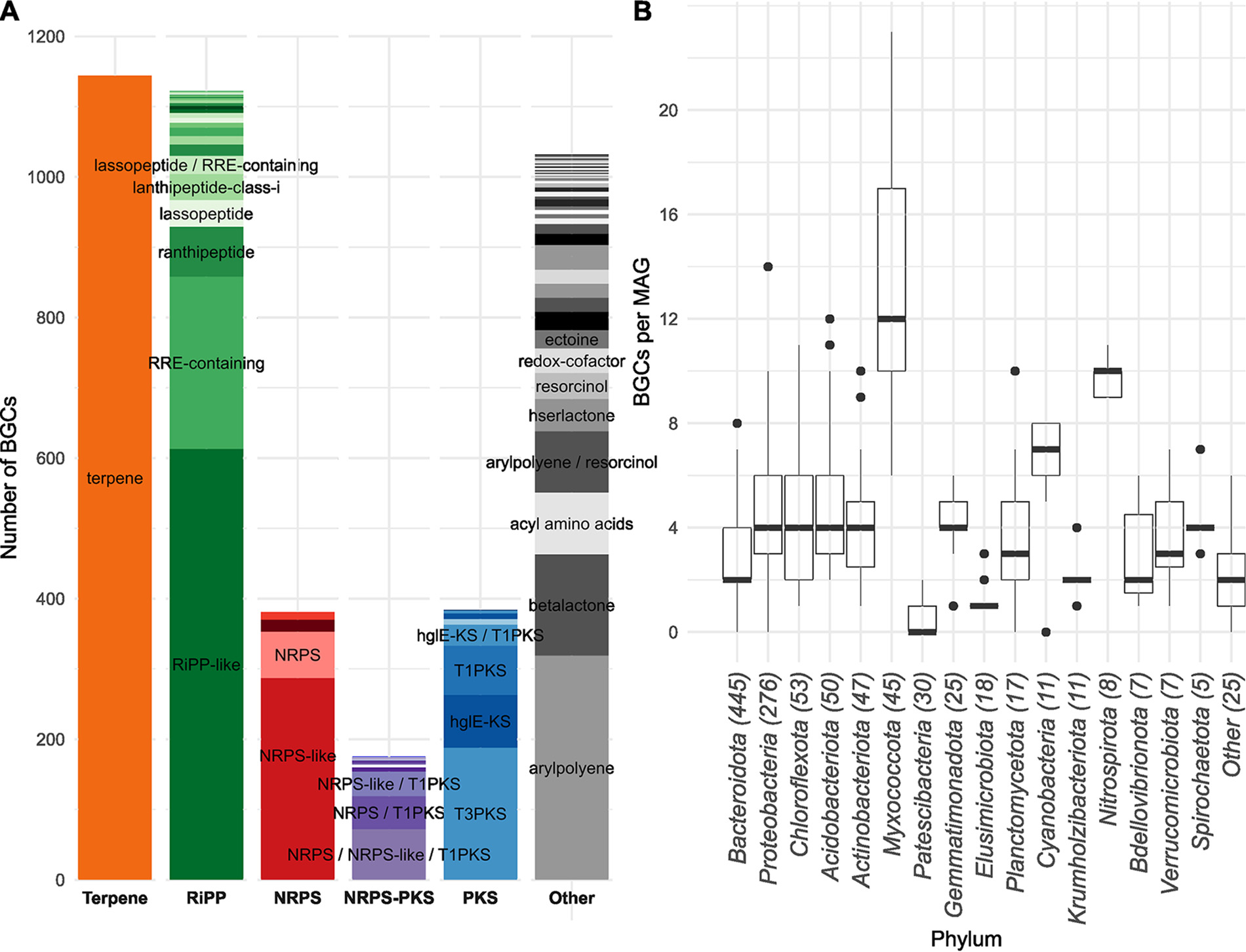
图1 高质量MAGs数据集中检测到的BGCs数量概述
2. 关键工艺细菌中的BGCs
在活性污泥中,属于丰富的和工艺关键型细菌的几个属显示出巨大而多样的生物合成潜力(图2)。丝状细菌可以引起泡沫,严重破坏废水处理效率。在常见且含量丰富的丝状细菌中,Candidatus Villigracilis和Ca. eofilum两个属含有萜烯,但几乎不含RiPPs。在硝化菌中,所有属均检测到RiPPs和萜类BGCs。在聚磷生物(PAOs)中,废水处理厂中数量最多的Ca. Phosphoribacter和Ca. Lutibacillus几乎没有生物合成潜力,而Dechloromonas和Ca. Accumulibacter则富含BGCs。这两个属在其生物合成潜力方面也表现出一定的可变性。总的来说,次级代谢物似乎与许多过程关键型细菌的生命策略有关,可能与生物膜或絮凝体形成、群体感应和微生物间竞争有关,但最终它们的功能完全未知。
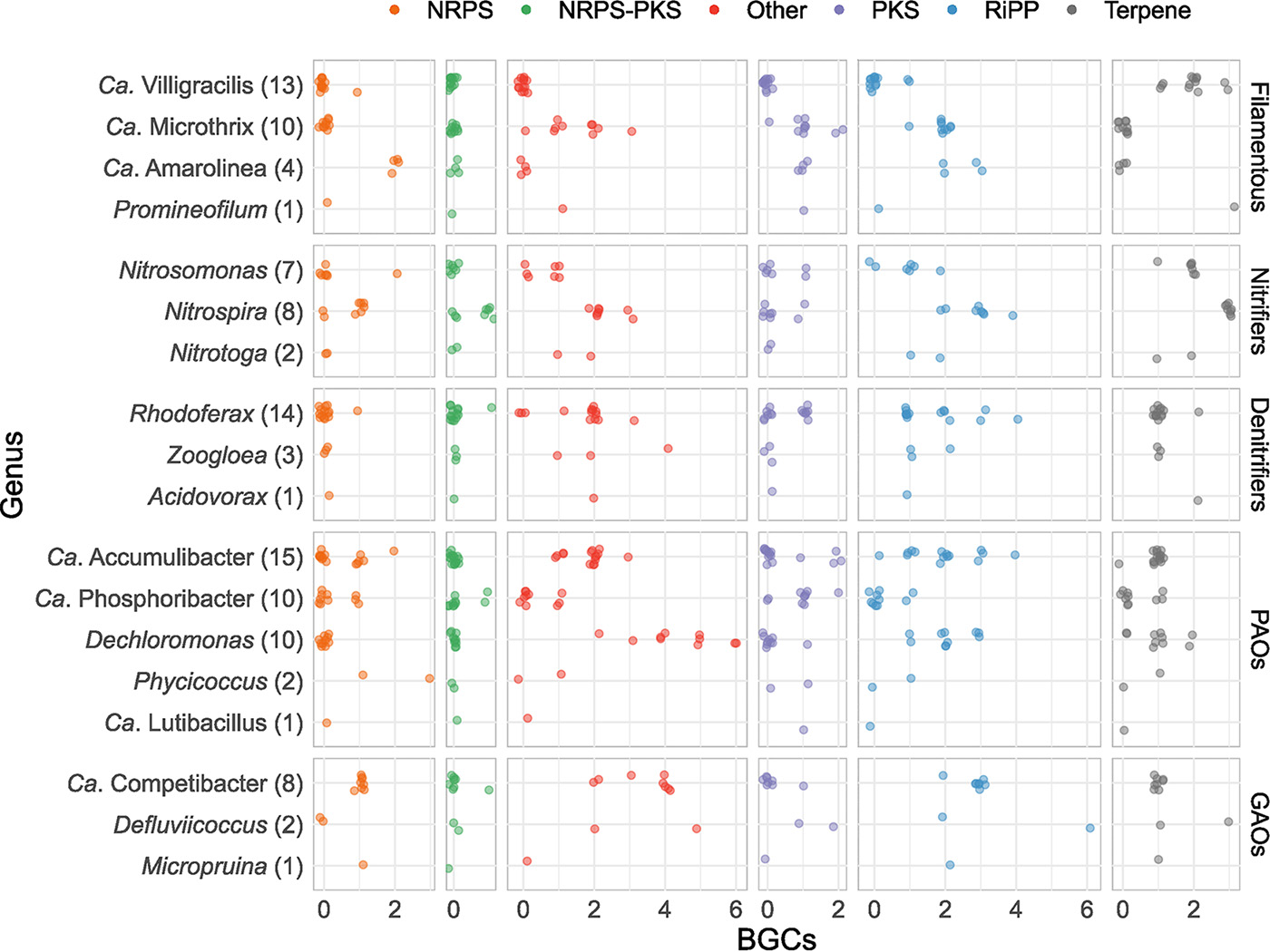
图2 所选属的BGCs在相关功能上和/或在养分去除的WWTPs中丰富
3. 硝化螺旋菌和粘菌中的BGCs
硝化螺旋菌和粘菌在AS环境中具有较高的BGC潜力和重要作用。在AS中,硝化螺菌属包括典型的亚硝酸氧化菌, N. defluvii和两个comammox物种-Ca. Nitrospira nitrosa和N. inopinata,它们的生物合成潜力尚未被研究过。为了评估在硝化螺菌中检测到的BGCs的新颖性和多样性,将我们的MAGs中的基因簇家族与NCBI RefSeq (大多数MAGs都是由短读数据生成的) 中可用的基因组进行了比较。所有硝化螺旋菌的生物合成潜力惊人地相似(图3)。在每个基因组中都检测到几个用于萜烯合成的BGCs,并且所有基因组都包含几个用于RiPPs的集群。Ca. Nitrospira nitrosa是唯一具有PK簇的种。
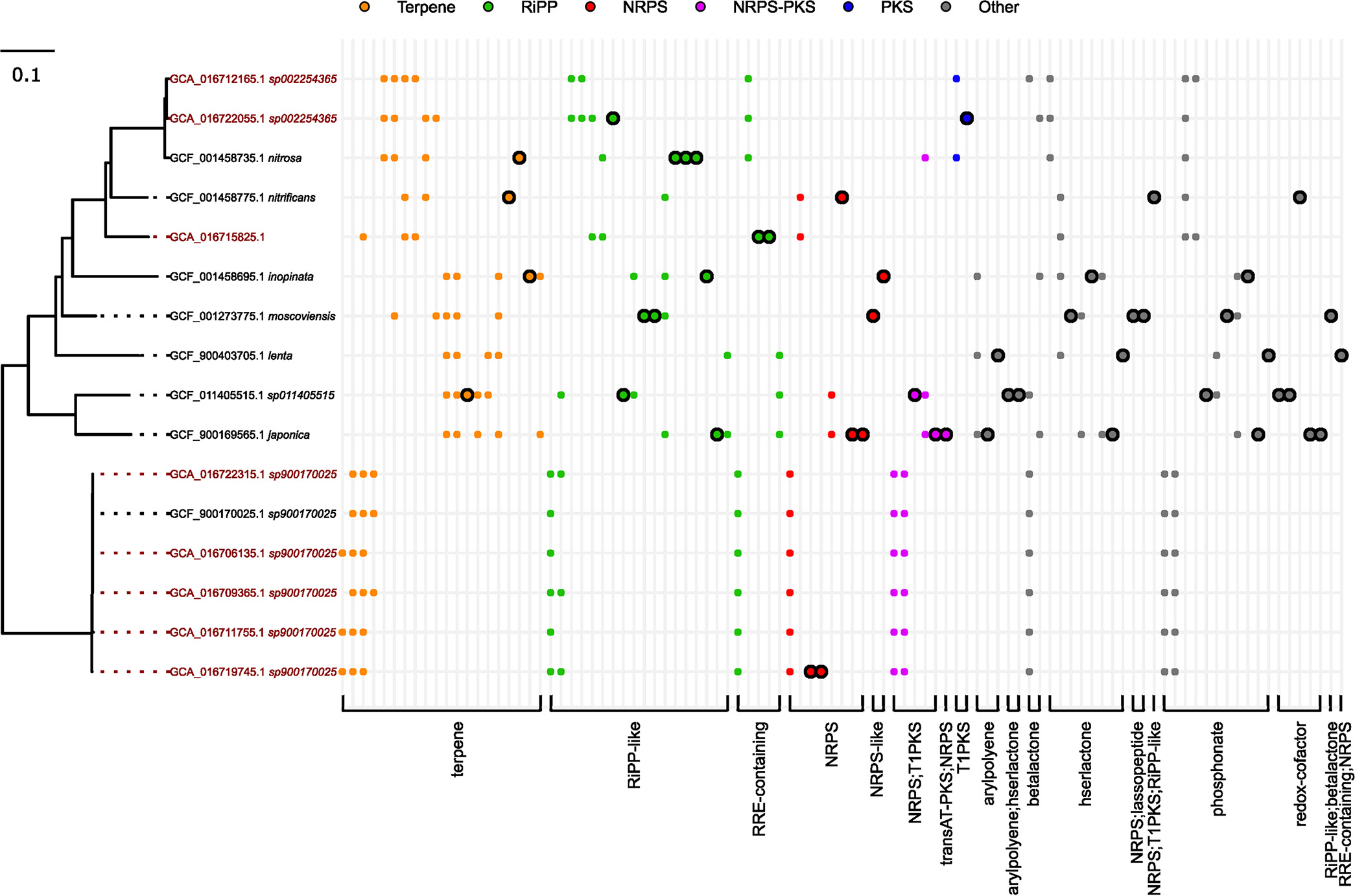
图3 硝化螺菌16个基因组,系统基因组树中基因簇家族的分布
选择Polyangiaceae多囊粘菌科与参考基因组的BGCs进行比较(图4)。多囊粘菌科包括Minicytis rosea,目前发现的拥有最大基因组(16 Mb)的细菌,其中包含47个BGCs。该科的10个MAGs中只有1个可以被分配到GTDB属,突出了这个家族在活性污泥系统中的新颖性。本研究的MAGs与所选参考基因组很少或没有共享BGCs。这表明从复杂的微生物群落中挖掘高质量MAGs,与从培养的代表菌中发现的基因簇家族相比,发现新的基因簇家族具有巨大的潜力。
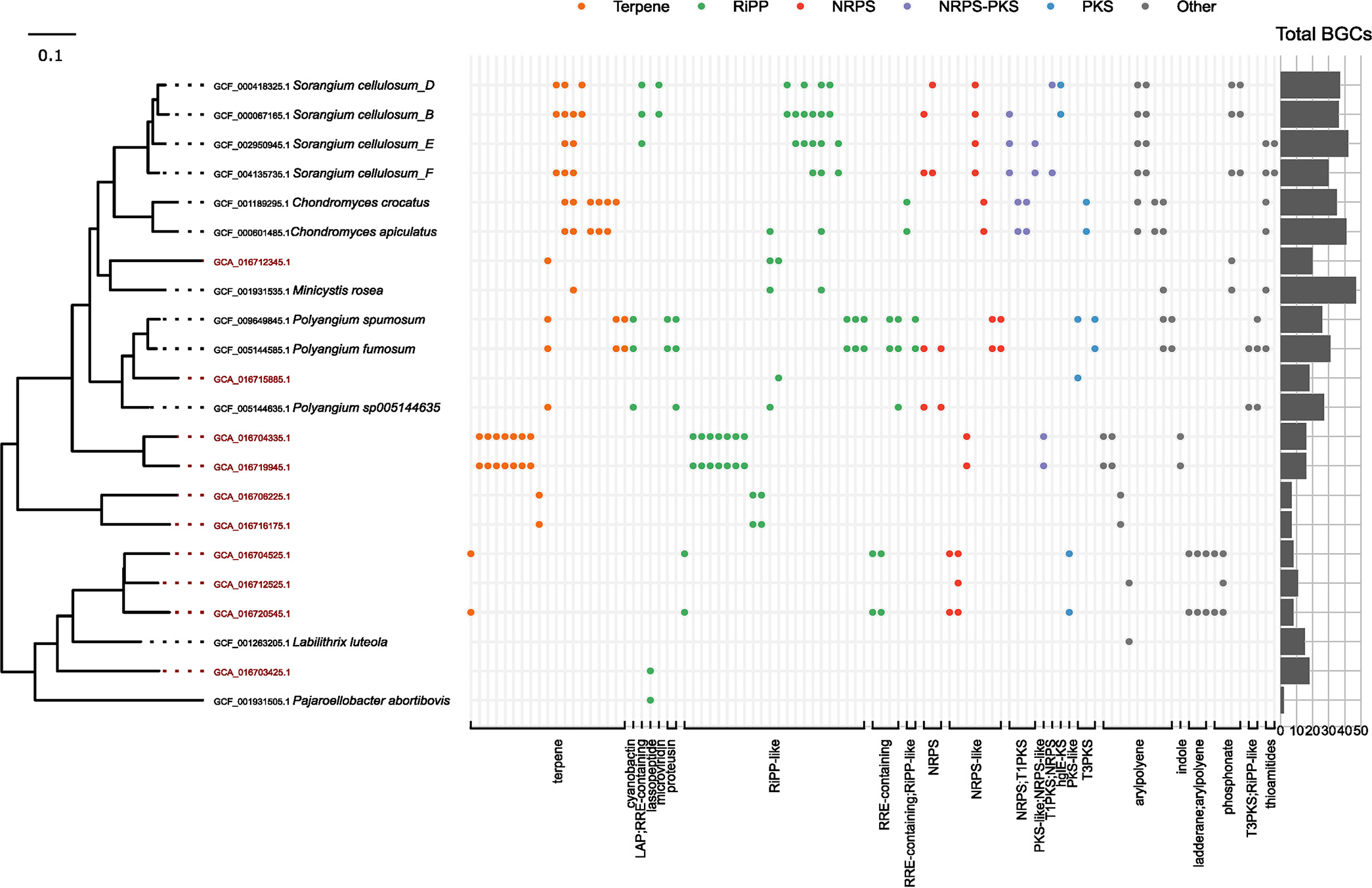
图4 多囊粘菌22个基因组,系统基因组树中基因簇家族的分布
4. 比较研究强调了从长读中获得高质量MAGs的必要性
为了评估不同的测序技术和生态系统对环境样本中BGCs恢复的影响,调查了五项在MAGs中挖掘BGCs的研究,并将其结果与本研究进行了比较。这些研究包括来自土壤、微生物垫的短读宏基因组数据和来自活性污泥和羊粪便的长读宏基因组数据。
对比显示,使用短读数据组装的MAGs来挖掘BGCs的结果大多是不完整的(图5)。在长读得到的HQ MAGs中,检测到大部分连续的BGCs。完整BGC比例的微小差异可能是由于使用了不同的长读技术(Nanopore以及PacBio),不同的测序深度和覆盖截断,或者基因组中模块化BGC类的比例差异。可以观察到与生态系统相关的BGCs回收比例的差异明显(图5)。NRP BGCs在土壤研究中更频繁地被检测到。在羊粪便的研究中,回收的BGCs大多为RiPPs。在所有研究中,检测到的多模块BGCs (NRPS、I型PKS和trans-AT PKS)非常短。在长读研究中,多模块BGCs的中位数仅为两个模块(图5),远短于大多数具有特征的此类BGCs。这表明在活性污泥和羊粪便生态系统中,模块化BGCs大多较短。
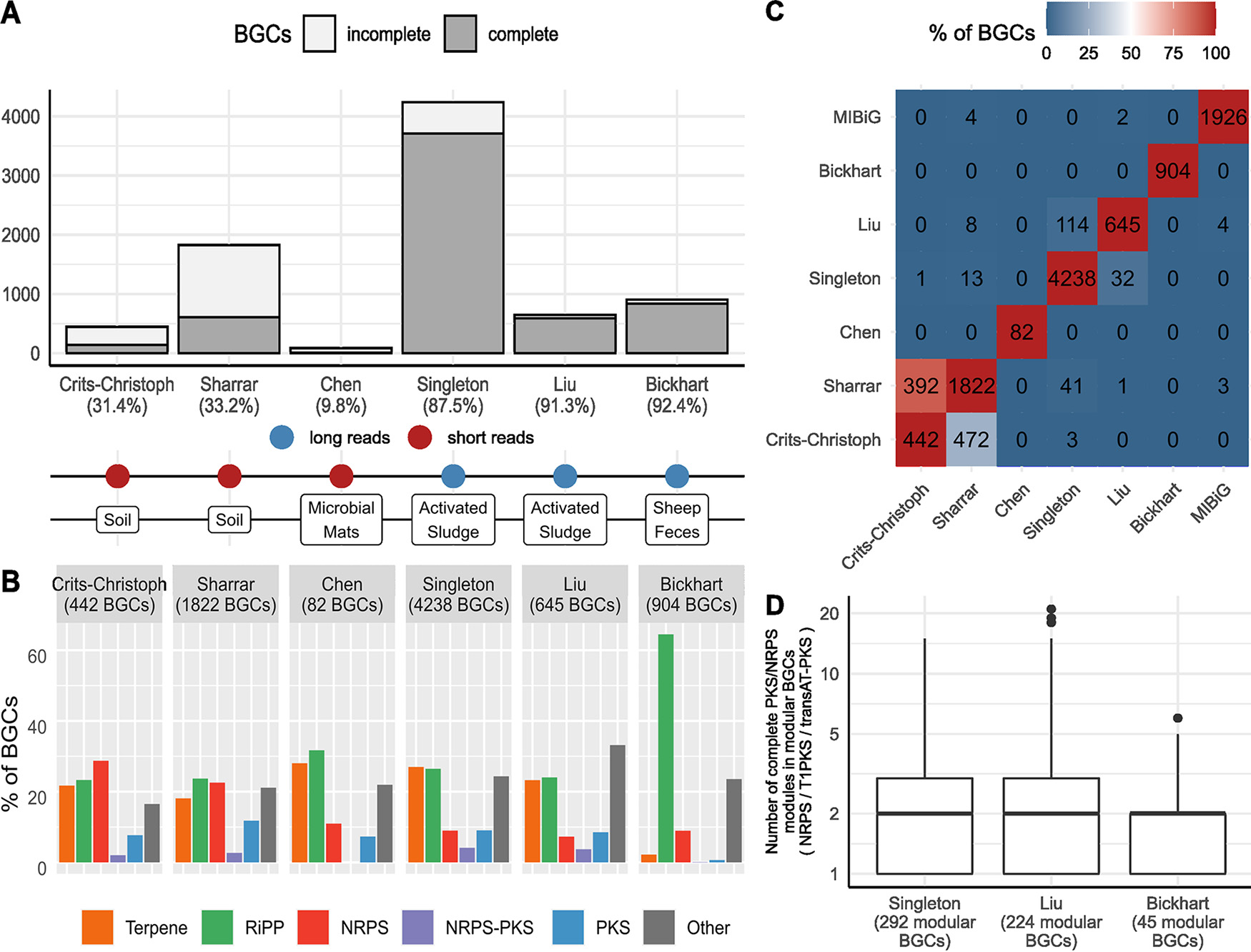
图5 高质量MAGs中BGCs挖掘研究比较
全文总结
活性污泥是世界范围内的重要资源,主要用于清洁水和保护人类和环境健康,但它在营养和水的回收及其对理想的循环经济的贡献方面也日益受到重视。负责活性污泥过程的复杂的、主要是未培养的微生物群落有大量的新BGCs,代表了未来表征的可获取来源。
从长读数据生成的高质量MAGs极大地提高了完整BGCs的回收率,促进了基因组挖掘,并为进一步研究提供了金标准基因组基础。从宏基因组中提取和表达BGCs的令人兴奋的进展表明了一种潜在的高通量产品表征方法,尽管产品检测仍然具有挑战性。应用宏转录组学来缩小原位高表达的潜在靶点可以增加成功的机会。
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



