NC项目文章 | BASALT-可利用二代及三代数据的宏基因组分箱及精炼工具
宏基因组分箱技术(Metagenomic binning)对于解析各种生态系统中未培养微生物的基因组至关重要,不仅可以有效地区分不同物种的序列,还能高效获取和分析单个不可培养菌株的基因组。但是现有的binning工具在有效获取metagenome-assembled genomes(MAGs)方面的效率不高,特别是对于高复杂度样品和低丰度微生物基因组,成为了一个极大的制约因素。比如对于土壤样品,基于目前的分箱组装方法只能利用到约30%的测序数据,对其它的高多样性环境样本的使用效率也普遍不超过50%。为了提高这一点,一种方法是进行混合组装,即结合短读和长读(short- and long-read,SRS & LRS) 宏基因组数据。然而,目前还没有工具能够在binning校正中同时整合SRS和LRS数据并补全基因组gaps。
近日,北京大学深圳研究生院余珂研究团队在Nature Communications上发表了研究论文“BASALT refines binning from metagenomic data and increases resolution of genome-resolved metagenomic analysis”。作者在文中开发了软件BASALT(Binning Across a Series of Assemblies Toolkit),可以同时结合二代和/或三代宏基因组序列进行高效的分箱和优化,实现对宏基因组测序数据的高效利用。BASALT软件可以免费在GitHub上获得(//github.com/EMBL-PKU/BASALT)。武汉AG代理基因参与该研究Nanopore宏基因组测序工作。
亮 点
BASALT拥有强大的处理宏基因组测序数据的能力。与目前的主流软件相比,无论是纯二代数据、二代+三代数据、或是纯三代数据(HiFi数据),BASALT处理产生的MAGs从数量和质量上都好于其他软件,其主要优势体现在:
可以同时输入多组装文件,包括合并组装(Co-assembly)的组装文件。利用多个样品产生多维度的contig序列覆盖度,可以更好地区分contig序列,减少杂合bins的产生。
整合了多个主流的分箱软件的分箱结果,利用核心算法Core sequences identification(CSI)找出组装后基因组的核心序列,进行去冗余、去污染、片段找回等一系列基因组优化步骤,可以显著提高MAGs质量和菌株水平的分辨率。
支持二代+三代数据,以及纯三代数据的binning。在三代数据存在时,其序列也会被应用于一系列优化步骤中,可以大大提高三代数据的利用效率。
支持多分箱软件的选择和多种数据类型的输入。用户可以:(1)依照喜好选择分箱软件;(2)选择使用完整的BASALT工作流程或部分功能;(3)输入组装文件(contigs或scaffolds)进行分箱和优化,或是输入已经完成分箱的bins仅进行优化。
结果
1. BASALT工作流程
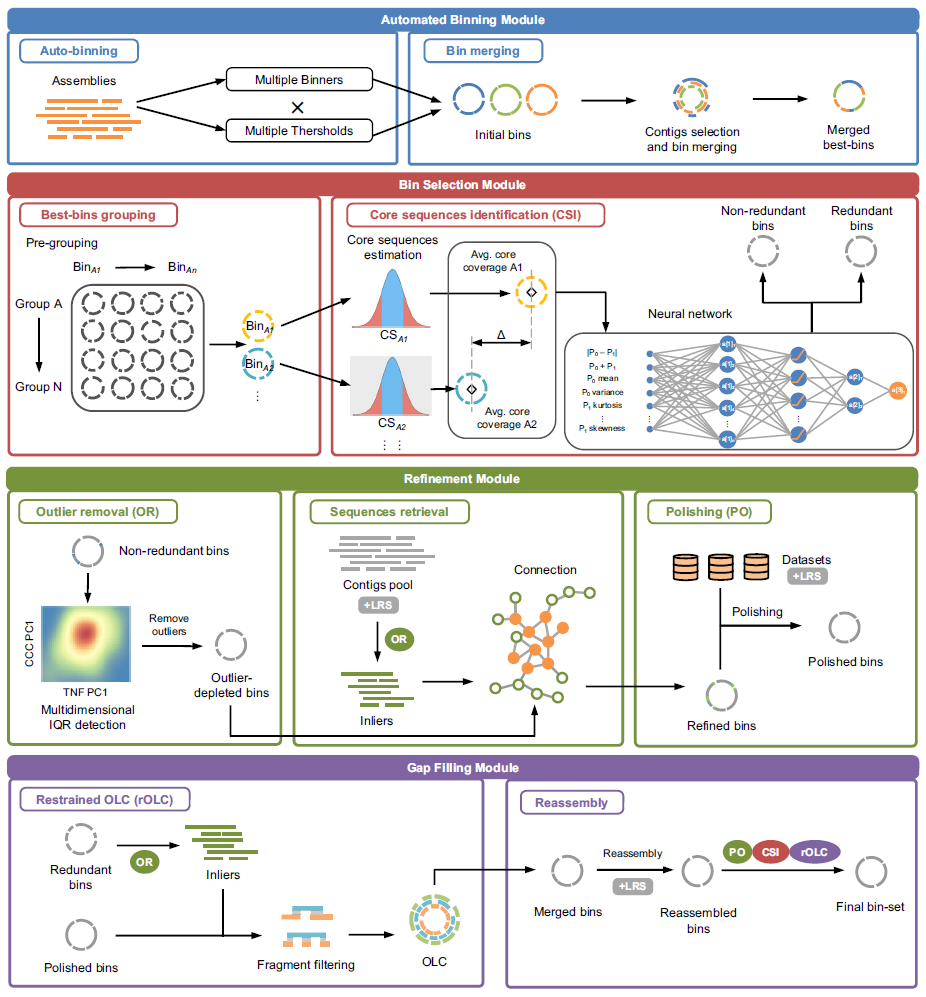
BASALT主要分为四个模块,包括自动分箱模块(蓝色)、基因组选择模块(红色)、优化模块(绿色)和空缺填补模块(紫色)
自动分箱模块:输入基于短序列(short-read sequences, SRS)或/和(long-read sequences, LRS)生成的单一组装文件、多组装文件和SRS+LRS混装文件进行自动分箱。利用多个分箱软件,在不同阈值下输出bins,并通过识别contig ID来合并相同的bins;
基因组选择模块:基于contig的覆盖率分布识别每个bin中的核心序列,通过神经网络识别bins之间覆盖度差值的阈值,帮助识别并去除冗余bins。
优化模块:异常序列去除(outlier removal, OR)功能通过识别bins的四碱基频率(TNF)以及核心覆盖系数(CCC)生成多维度图谱,移除bin中的潜在污染序列。然后,序列召回(sequences retrieval)功能将剩下未分箱的序列(包括三代序列)通过双端追踪法(pair-end tracking)选择性填补bins中的空缺,包括找回多拷贝基因。下一步中,在三代数据存在的情况下,将对每个bin中的LRS进行单独修正。
空缺填补模块:为了进一步提高bins的质量,空缺填补模块首先回收冗余基因组序列,利用限制性重叠-布局-共识法(restrained overlap-layout-consensus, rOLC)对序列进行填补。随后,重组装功能对填补后的序列进行重组装(在二+三代序列存在的情况下,将使用混装工具)。最后,经过一系列的修正、去冗余等finalisation工作后,生成最终的bins。
2. 利用CAMI数据集评估BASALT的性能
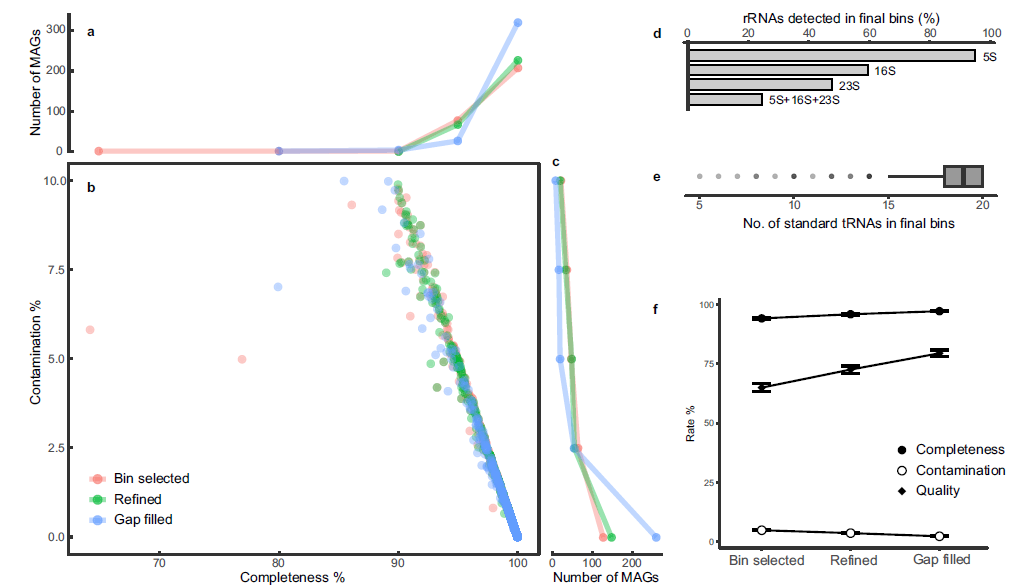
评估采用Critical Assessment of Metagenome Interpretation (CAMI)中的CAMI-high数据(共596的标准基因组)。使用二代+三代数据混装产生的组装文件,BASALT可以恢复其中的392个满足完整度 ≥ 50,污染度 ≤ 10的MAGs。经过基因组选择、优化和空缺填补每一个模块处理后,所产生的MAGs无论从完整度(Completeness)、污染度(Contamination)和总质量(完整度-5*污染度)上都有很大的提升(图a, b, c, f)。
3. BASALT与其他主流软件的比较
评估同样选用CAMI-high数据集混装产生的组装文件。MAGs的评估采用更严格的质量值(完整度 - 5*污染度 )≥ 50为标准。在利用VAMB(紫色)、DASTool(绿色)、
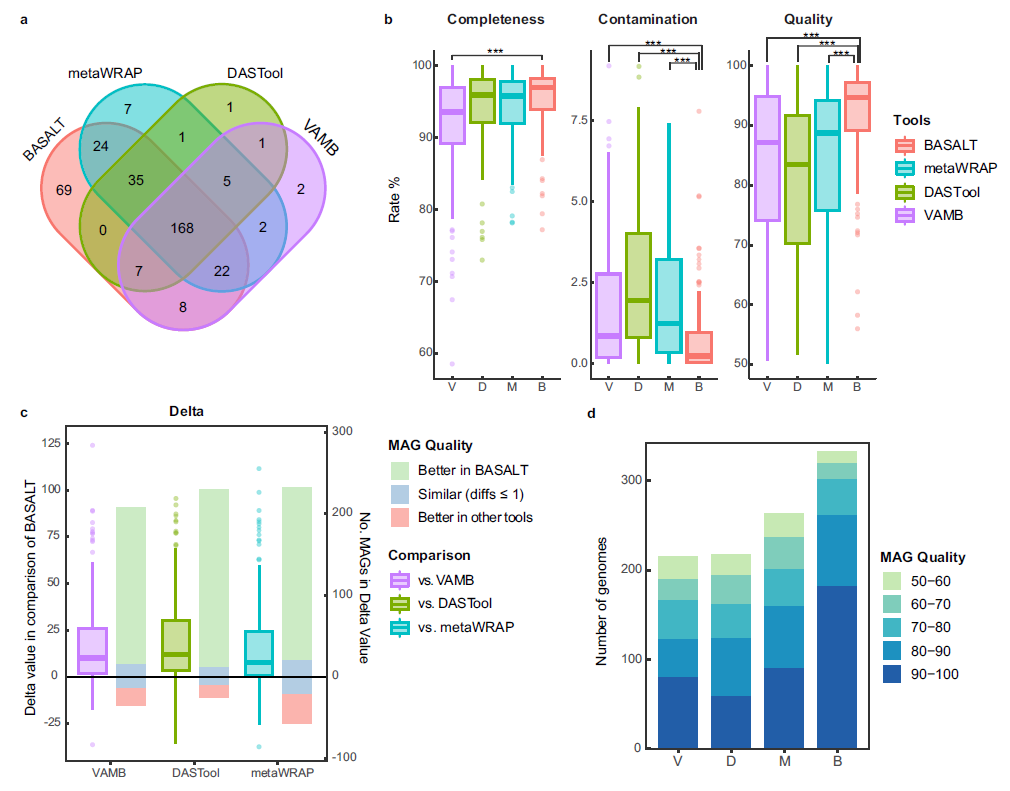
metaWRAP(青色)和BASALT(红色)四个软件处理后,对比发现BASALT比VAMB、DASTool和metaWRAP分别多63.7%、61.5%和33.3%的MAGs,其中包括独有的69个MAGs(图a)。在四个软件共同获得的168个MAGs中,BASALT获得的基因组质量也同样显著高于其他软件(图b)。两两比较的结果显示,BASALT生成的MAGs质量普遍高于其他软件,其质量更好的MAGs数量对比VAMB、DASTool和metaWRAP分别高出约9.6倍、14.6倍和6.1倍(图c)。这使得BASALT与VAMB、DASTool和metaWRAP相比,质量值大于90(完整度 - 5*污染度 ≥90)的MAGs,BASALT获得的数量分别是2.28、3.59和2.02倍(图d)。
4. BASALT在真实数据集上的评估
除测试数据集外,作者团队还选用了十个真实数据集,包括SRS,SRS+LRS,以及LRS(HiFi)数据,涵盖了盐湖沉积物(sediment)、人类肠道、海水(marine)、南极土壤(soil)、活性污泥(activated sludge, AS)、鸡肠道、羊肠道、热泉、厌氧反应器(anaerobic digester, AD)等多种环境类型。通过与metaWRAP(SRS或SRS+LRS数据)和MAG-HiFi-Pipeline(LRS HiFi数据)比较,BASALT所获得的MAGs均多于其他软件,特别是高质量基因组(完整度 ≥ 90,污染度 ≤ 5)的数量有较大的提升。
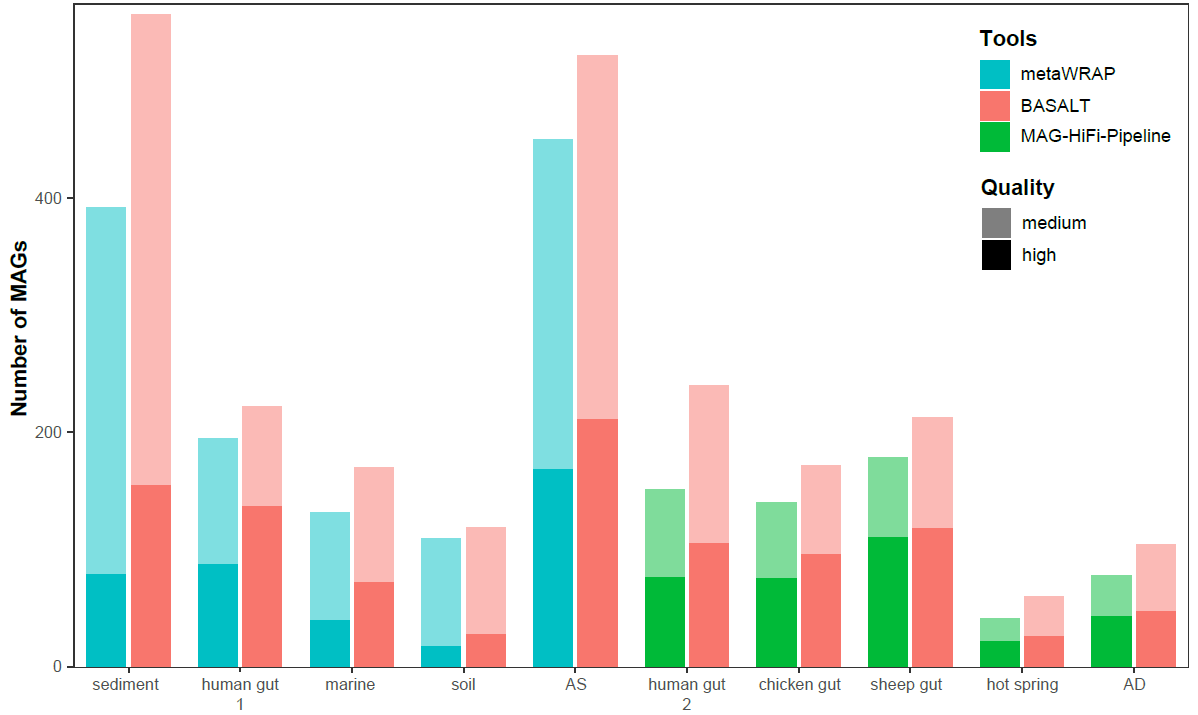
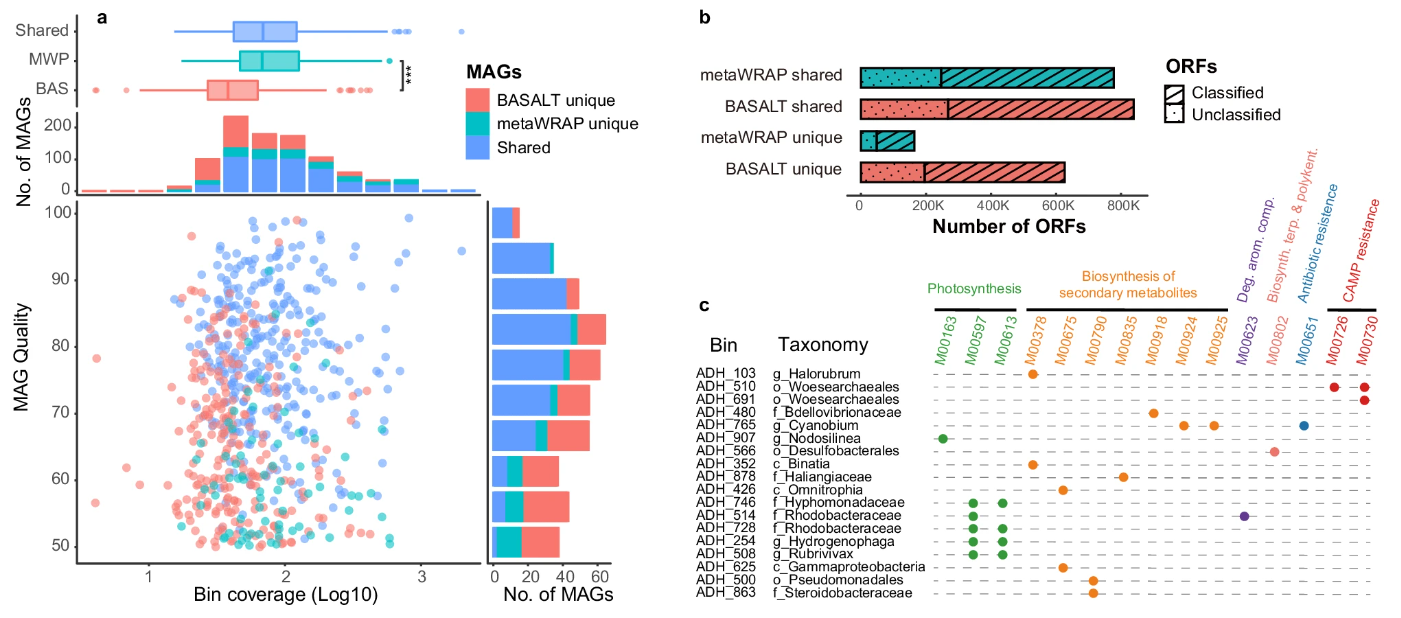
以高度复杂度的盐湖沉积物微生物群落样品为例(纯二代分析数据), BASALT可以比metaWRAP多获得30%的MAGs,对比发现,BASALT在恢复低丰度的MAGs上比metaWRAP更有优势(图a)。在功能注释分析中,BASALT可以比metaWRAP多获得47.6%的非冗余开放式阅读框(ORF)序列(图b),使得BASALT恢复的MAGs在功能模块上更为完整(图c)。以上结果表明,BASALT能够从测序数据中获得高分辨率、高质量的基因组,更深层次地挖掘了以往工具未能获得的微生物信息。
5. BASALT识别其他工具无法获得的微生物分支
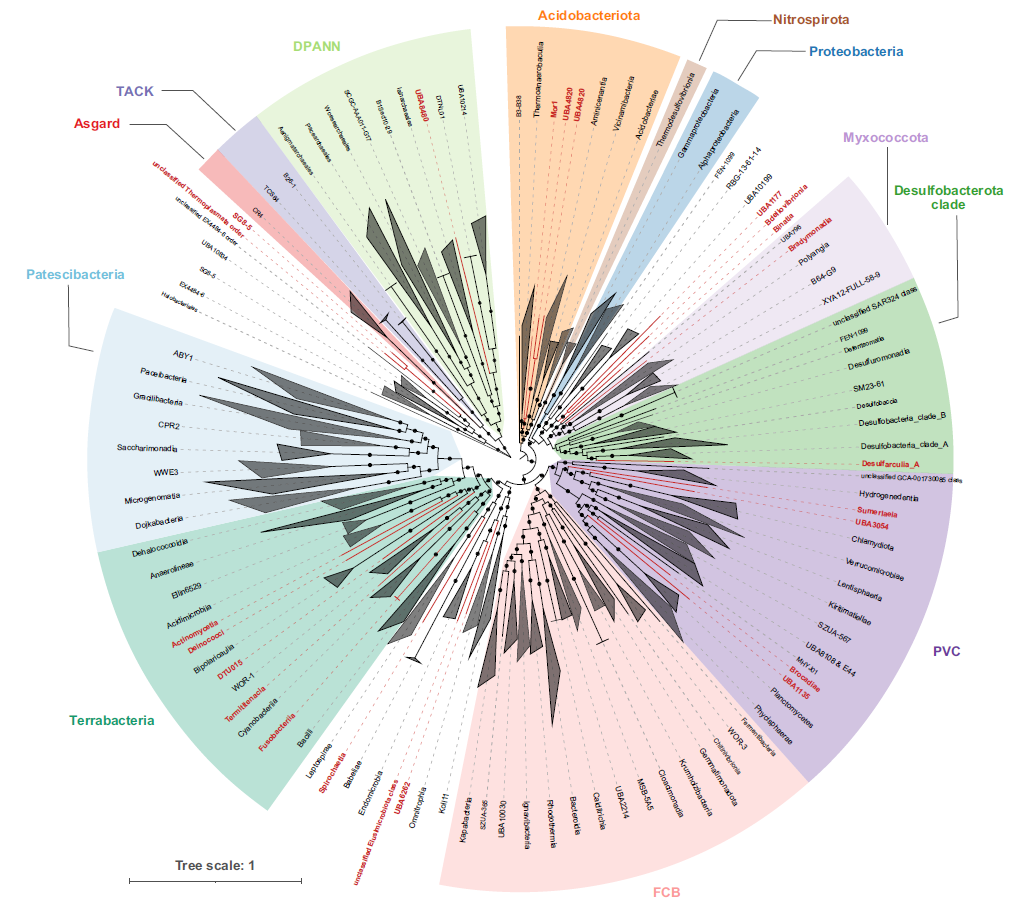
从盐湖沉积物样品中,BASALT共获得557个MAGs,共涵盖54个门;而metaWRAP识别了392个MAGs,涵盖45个门,这些门都被BASALT检测到。BASALT独特地识别出了metaWRAP未发现的9个门,包括21个纲水平上的细菌分支和2个目水平上的古菌分支(红色高亮分支)。这些独特的分支有助于我们发掘复杂环境中未被探索的微生物及功能元件。例如,在盐湖沉积物中,研究团队发现了Nanoarchaeota门的两个古菌MAGs中有抗抗菌肽(CAMP)功能模块;而在Asgardarchaeota门下归类为Prometheoarchaeum属的MAGs之前主要在海洋样本中报道,本文首次报道其在内陆盐湖中存在。
总之,BASALT相较其他主流软件,无论从获得基因组的质量和数量上均有显著优势,而这一优势在高微生物复杂度的样本中更加明显。利用BASALT分箱,可以更深层次地挖掘了以往工具未能获得的微生物信息。
第一作者:仇知光、余珂
通讯作者:余珂

仇知光
博士,北京大学深圳研究生院,任特聘副研究员。主要从事环境微生物组的研究,研究方向为极端环境微生物的功能挖掘与微生物进化。利用生物信息学技术,在微生物生态、功能探索、资源挖掘与应用等方面取得一系列成果。发表论文30余篇,包括以第一或通讯作者在Nature Communications, Biotechnology Advances, Environmental Science & Ecotechnology等国内外相关领域高水平期刊上发表的多篇论文。

余珂
博士,北京大学深圳研究生院,任助理教授/研究员,环境科学与工程学、计算机应用技术双学科博导。主要从事环境微生物组及生物信息学方法研究,专注于生物信息学分析算法、流程及可视化开发,结合培养组发掘极端环境体系中具有应用潜力的微生物。研究同时关注于多宏组学联用技术开发,解析群落微生物的生态位分异及其互作关系。成果包括论文80余篇,含以第一作者或通讯作者身份在包括Nature Communications, Microbiome, Environmental Science & Technology,Water Research等微生物组学、环境工程等多领域多个期刊的多篇论文。



 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



