Science 6连发 | 人类T2T基因组及后续研究

前言
1990年,人类基因组计划(Human Genome Project, HGP)正式启动,随着测序技术的进步,人类基因组也不断在更新和完善,但是一直以来,约8%的基因组尚未被测序和破解,在这些区域中,多数为高度重复序列,无法通过常规的测序获得这部分的序列。
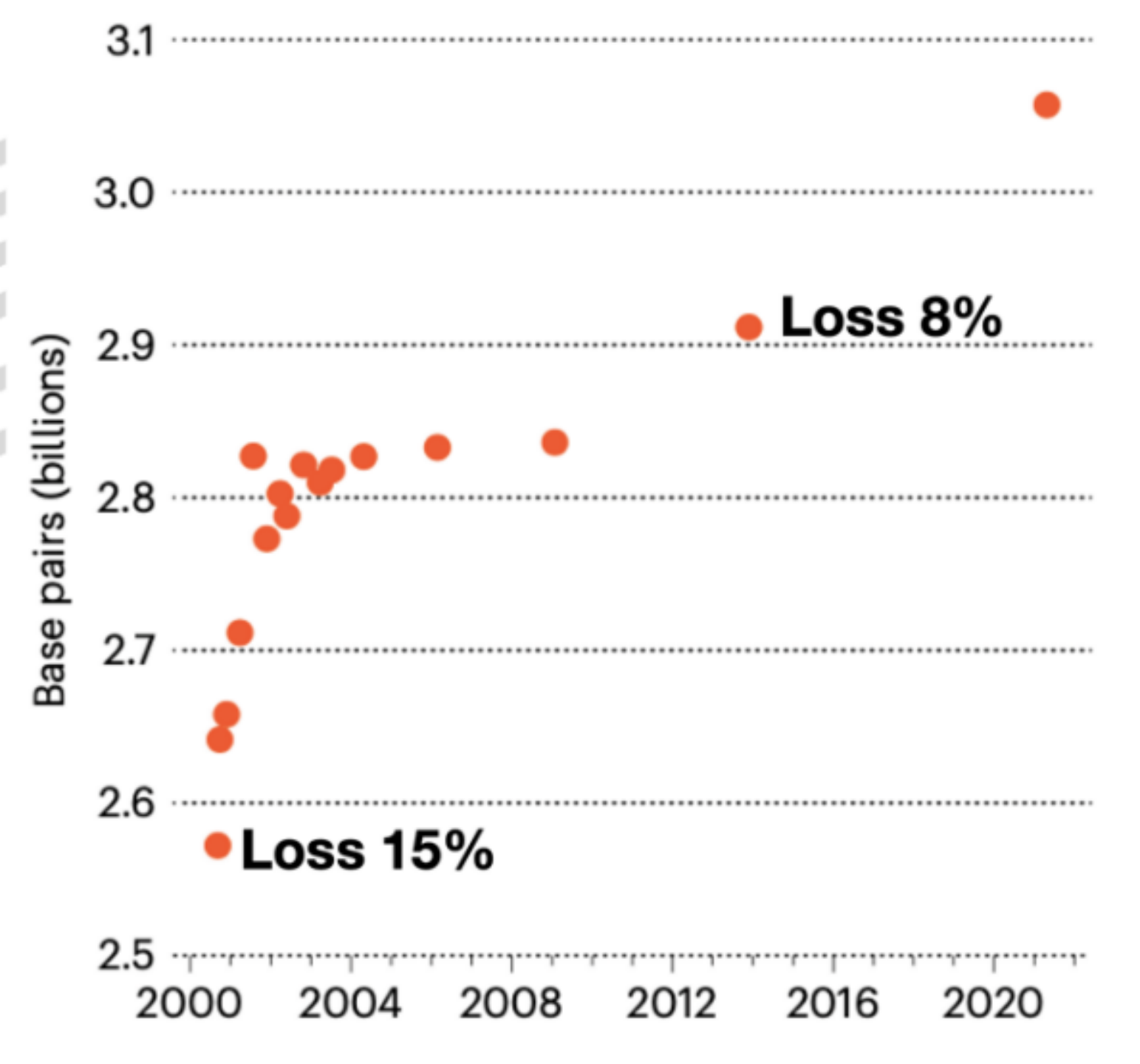
图1 人类基因组组装历史进程
2022 年初,Nature 杂志对「可能在未来一年对科学产生影响」的 7 项技术进行了展望。其中,T2T基因组位居 7 大技术展望之首。
长读长测序技术是绘制人类T2T基因组的必备工具。其中,Oxford Nanopore(牛津纳米孔)测序技术可以一次读取长达 100 万个碱基的序列;PacBio 公司开发的 HiFi 测序技术可以读取超过 20Kb,单碱基准确度达 99.9%。
T2T联盟结合了Nanopore超长及PacBio测序技术,利用两者的优势互补,构建了人类完整基因组T2T-CHM13,并于2022年4月1日,以6篇研究论文连发的形式,在Science期刊汇报人类T2T基因组的研究成果,首次测序并破译了完整的人类基因组,并基于T2T-CHM13进行了后续深入研究。
AG代理基因作为国内首批引入Nanopore测序平台的科技公司,专注于Nanopore超长测序技术的研发和生产,并已实现Nanopore超长序列(N50>100kb)的稳定产出,为广大科研学者提供T2T基因组测序和研究服务。
6篇Science论文及研究概要
01
人类完整基因组
(The complete sequence of a human genome)

研究概要:
自2000年首次发布人类参考基因组以来,人类参考基因组只覆盖了基因组的常染色质部分,留下了关键的异染色质区域未被破解。T2T联盟解决了此前未被组装的剩余8%基因组,并提供了一个完整的,含有30.55亿碱基对的人类完整基因组(T2T-CHM13),包含除Y染色体以外所有染色体的无gap组装,同时纠正了之前参考基因组中的组装错误。T2T-CHM13中新引入了近2亿个碱基对序列,包含1956个新基因。完成的区域包括所有的着丝粒卫星阵列,片段重复,以及所有染色体的短臂,解锁这些基因组的复杂区域,为后续进行变异和功能研究提供重要的研究基础。
02
人类着丝粒的基因组和表观遗传图谱
(Complete genomic and epigenetic maps of human centromeres)

研究概要:
基于人类完成的T2T基因组(T2T-CHM13),发现了着丝粒重复区域变异和进化的新模式,比较了不同个体X染色体着丝粒,并揭示了着丝粒区域的结构、表观遗传和序列变化。该研究指出,对着丝粒区域进行表观遗传和遗传变异研究,能够更好地确定和研究着丝粒表观基因的定位和遗传趋势。
03
完整的参考基因组提升了对人类遗传变异的分析
(A complete reference genome improves analysis of human genetic variation)

研究概要:
该研究通过将T2T-CHM13与人类基因组历史版本(GRCh38)进行比较,发现CHM13基因组增加了近200 Mb的序列,纠正了历史版本数千个组装错误,并为临床医学和功能研究解锁了人类基因组中最复杂的区域。该研究分别用3,202个短读数据样本和17个长读数据样本进行read比对和变异检测,分析并指出新的T2T-CHM13基因组大幅改进了人类遗传变异分析。
04
人类基因组的表观遗传图谱
(Epigenetic patterns in a complete human genome)

研究概要:
该研究利用Nanopore直接测序优势,构建完整的人类基因组甲基化图谱,与WGBS方法相比,基因组覆盖度增加了32.8M,并能够对更多CpGs(10%3.18M)进行研究。该研究针对高度同源的基因家族及串联重复进行研究,发现高度相似的序列间存在表观遗传和转录活性的差异,突显了局部染色体环境作为表观遗传调节因子的重要性。T2T-CHM13对表观遗传图谱,是重大的生物学进展,为利用ONT长读测序进行物种表观遗传研究奠定了基础。
05
完整的人类基因组中的片段复制及其变异
(Segmental duplications and their variation in a complete human genome)

研究概要:
研究人员将完整的人类T2T基因组(CHM13)与人类参考基因组(GRCh38)进行比较发现,片段重复(Segmental duplications,SDs)占到新增序列的三分之一。通过与其他人类和非人灵长类物种组装进行比较,发现SDs区域SNV密度显著提升,同时发现SD区块通常作为一个整体被甲基化或非甲基化,其中存在许多候选基因的串联重复或大的散在重复。该研究使用T2T-CHM13基因组系统地重建并揭示了前所未有的结构杂合性模式,以及人类及其近亲在SD结构中前所未有结构杂合度模式和巨大的进化差异。
06
从端粒到端粒:人类重复元素的转录和表观遗传状态
(From telomere to telomere: The transcriptional and epigenetic state of human repeat elements)

研究概要:
该研究基于T2T-CHM13实现了一个全面的重复注释流程,并利用这种方法,更新了的人类重复序列信息,并改进了以前的重复注释。在T2T-CHM13中,发现了43个以前未知的重复和重复变体,并确定了19个复杂、复合的重复结构,这些结构通常携带功能基因。利用ONT测序数据生成的PRO-seq和CpG甲基化数据,揭示了新转录本、序列差异、CpG之间的相关性。本文所述的全面重复注释可作为扩充人类基因组序列的重要资源,并揭示特定重复对人类基因组的影响。
总结
人类T2T基因组及后续研究,标志着基因组研究4.0时代正式到来。
AG代理基因结合多篇文献报道及项目组装实例,针对T2T基因组推出以下测序策略:

以物种T2T基因组为研究基础,为各位研究学者总结后续研究方向:

有科研需求的老师,请联系AG代理基因(Tel: 027-62435310; service@speedracings.com)或在后台留言,AG代理基因基因组事业部将为您专业定制T2T基因组研究策略及研究方案。
参考文献:
[1] Sergey Nurk et al., (2022) The complete sequence of a human genome. Science. Doi: 10.1126/science.abj6987
[2] Sergey Aganezov et al., (2022) A complete reference genome improves analysis of human genetic variation. Science DOI: 10.1126/science.abl3533
[3] Mitchell R. Vollger et al., (2022) Segmental duplications and their variation in a complete human genome. Science DOI: 10.1126/science.abj6965
[4] Complete genomic and epigenetic maps of human centromeres. Science(2022), DOI: 0.1126/science.abl4178
[5] A. Gershman et al., Epigenetic patterns in a complete human genome. Science 376, eabj5089 (2022). DOI: 10.1126/science.abj5089
[6] S. J. Hoyt et al., (2022) From telomere to telomere: The transcriptional and epigenetic state of human repeat elements Science DOI: 10.1126/science.abk3112
 027-62435310 |
027-62435310 |
 service@speedracings.com |
service@speedracings.com |



